1.propiedades fisicoquimicas del dna
- 1. DNADNA
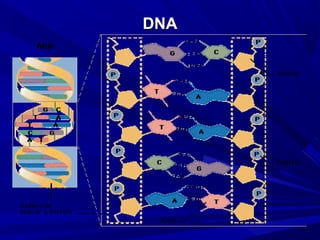
- 2. DNA ESTRUCTURADNA ESTRUCTURA LALA ESTRUCTURAESTRUCTURA DEL ADN ESTADEL ADN ESTA DEFINIDA PORDEFINIDA POR LA SECUENCIALA SECUENCIA DE LAS BASESDE LAS BASES NITROGENADANITROGENADA S EN LAS EN LA CADENA DECADENA DE NUCLEOTIDOSNUCLEOTIDOS
- 3. ESTRUCTURA DE BASES NITROGENADASESTRUCTURA DE BASES NITROGENADAS DEL DNA Y RNADEL DNA Y RNA
- 5. A G C T Hombre, H.sapiens 0.29 0.18 0.18 0.31 Bovino, Bos taurus 0.26 0.24 0.23 0.27 Levadura, S.cerevisiae 0.30 0.18 0.15 0.29 Mycobacterium sp. 0.12 0.28 0.26 0.11 Composición en bases del DNA en algunas especies
- 6. 1. La relación purinas/pirimidinas es igual a 1 Es decir, A+G = C+T 2. En todos los DNA estudiados, la proporción molar de A es igual a la de T, y la de G igual a la de C. Es decir, A = T y G = C Reglas de Chargaff
- 7. 1. El DNA es una doble hélice plectonémica y dextrógira, con un paso de rosca de 3.4 nm 3.4 nm Modelo de Watson - Crick, A
- 8. Modelo de Watson-Crick, B 2. Cada una de las dos hélices es un polinucleótido entrelazado con el otro de manera que su polaridad es opuesta (es decir, corren en sentido antiparalelo) 5’ 3’ 5’ 3’
- 9. 3. El eje ribosa-fosfato se sitúa hacia el exterior de la doble hélice, en contacto con el solvente 4. Mientras que las bases nitrogenadas (anillos planares) se sitúan, apiladas, hacia el interior de la estructura, en un entorno hidrofóbico Modelo de Watson-Crick, C
- 10. 5. Las bases están situadas en planos aproximadamente perpendiculares al eje mayor de la doble hélice. La distancia entre planos es de 0.34 nm Modelo de Watson-Crick, D 0.34 nm
- 11. Modelo de Watson-Crick, E N N N N N HH N N O O CH3 H A T 6. Cada base interacciona con su opuesta a través de enlaces de hidrógeno, y de manera que: (a) Adenina (A) sólo puede interaccionar con timina (T) (y viceversa), a través de dos puentes de hidrógeno, y
- 12. N N N N O H N H H N N O N H H G C (b) Guanina (G) sólo puede interaccionar con citosina (C) (y viceversa), a través de tres puentes de hidrógeno
- 13. 3’ 2’ 1’ 5’ 4’ 7. La base está situada en posición anti-8. La desoxirribosa en forma furanósica 9. El anillo furanósico está en conformación endo-2’ Modelo de Watson-Crick, F
- 14. 10. El eje de la doble hélice no pasa por el centro geométrico del par de bases. Esto determina que la hélice presente un surco ancho y un surco estrechoSurco ancho Surco estrecho Modelo de Watson-Crick, G
- 15. Paso de rosca 3.4 nm Distancia entre 0.34 nm planos de bases Pares de bases/vuelta 10 Anchura 2.4 nm Geometría de la doble hélice (DNA-B) 0.34 3.4 2.4
- 16. Interacciones débiles que mantienen la estructura del DNA 1. Enlaces de hidrógeno entre bases complementarias 2. Interacciones hidrofóbicas entre planos de bases contiguos (int. de apilamiento, stacking) 3. Interacciones iónicas del fosfato con moléculas electropositivas (histonas, poliaminas, etc.)
- 17. 1. Compatible con los datos cristalográficos 2. El modelo predice una determinada densidad lineal que se cumple para todos los DNAs conocidos 3. El DNA rico en GC debe ser más difícil de desnaturalizar que el rico en AT. Esta predicción se cumple perfectamente. 4. El estudio de frecuencias de vecino más próximo (A.Kornberg) sólo es compatible con un modelo complementario y antiparalelo p.e.: el par AG tiene la misma frecuencia que el par CT; AT tiene la misma frecuencia que TA, y así sucesivamente. Pruebas experimentales de la estructura del DNA
- 18. A B Z Grosor 2.6 2.4 1.8 Giro Dextro Dextro Levo Bases/vuelta 11 10.4 12 P.de rosca 2.5 3.4 4.5 Inclinación 19º 1º 9º plano bases Distintas formas del DNA
- 19. DNA-A 1.Doble hélice plectonémica y dextró- gira 2. Planos de bases oblicuos respecto al eje de la doble hélica 3. Propio de RNAs en doble hélice, o de híbridos DNA-RNA 4. Más ancha y corta que DNA-B
- 20. DNA-Z 1. Doble hélice plectonémica y levógira 2. Zonas de secuencia alternante -GCGC- 3. Conformación de G es syn- en lugar de anti- 4. Más estrecha y larga que DNA-B
- 22. Desnaturalización del DNA T, ºC % Incremento Absorbancia a 260 nm La desnaturalización térmica del DNA sigue una curva sigmoide. El punto medio, Tm, está relacionado con el conte- nido en G+C. Así, la muestra B tiene un mayor contenido en G+C que A.
- 23. La temperatura de fusión (Tm) necesaria para desnaturalizar la mitad del ADN de una mezcla (punto medio de la reacción ADN doble hélice → ADN hélice sencilla) esta directamente relacionada con el contenido en G+C, a mayor contenido en G+C mayor temperatura de fusión (Tm).
- 24. El estado físico de los ácidos nucleicos está relacionado con su capacidad de absorción de la luz ultravioleta (UV) a 2.600 . El menor grado de absorción se produce en estado de doble hélice, la absorción aumenta cuando se produce la desnaturalización pasando a estado de hélice sencilla (efecto hipercrómico, aumento de la absorbancia) y, por último, si degradamos este ADN de hélice sencilla a nivel de nucleótidos libres, de nuevo aumenta la absorbancia. Efecto Hipo crómico del DNA
- 26. ¿Para qué purificar DNA?¿Para qué purificar DNA? Aplicaciones
- 27. AplicacionesAplicaciones ForenseForense Dx enfermedades infecciosasDx enfermedades infecciosas Dx enfermedades congénitasDx enfermedades congénitas Clonación de GenesClonación de Genes GenómicaGenómica Control de calidad microbiológicoControl de calidad microbiológico BiodiversidadBiodiversidad Identificación de especiesIdentificación de especies
- 28. ¿Cómo purificar DNA?¿Cómo purificar DNA?
- 29. Los 3 pasos básicos para purificar DNALos 3 pasos básicos para purificar DNA Lisis celular Fraccionamiento de los ácidos nucleicos Concentración de los ácidos nucleicos 1.1. 2.2. 3.3.
- 30. 1. Lisis celular1. Lisis celular Lisis enzimática de tejidos animales Tampón de lisis con detergente. Algunos protocolos Con proteinasa K. Lisis de bacterias o levaduras. Lisozima (Bacterias Gram-positivas) liticasa (levaduras). Detergente y pH alcalino para plásmidos. Homogenizadores mecánicos Agitador mecánico, a la muestra se adicionan esferas. Congelación descongelación Se usa nitrógeno liquido.
- 31. 2. Fraccionamiento de los ácidos nucleicos2. Fraccionamiento de los ácidos nucleicos Preparación de lisados crudos No se purifica Salting-out methods Se precipitan las proteinas y contaminantes con sales de acetato de amonio o potasio Fraccionamiento con solventes orgánicos Fenol/cloroformo/alcohol isoamílico
- 32. 3. Concentración de ácidos nucleicos3. Concentración de ácidos nucleicos Precipitación con: Etanol Isopropanol En presencia de sales
- 33. 2. SEPARACION DE FRAGMENTOS DE2. SEPARACION DE FRAGMENTOS DE DNADNA Electroforesis
- 34. ¿Cómo hacer una electroforesis de¿Cómo hacer una electroforesis de DNA?DNA?
- 35. Electroforesis de DNAElectroforesis de DNA
- 36. Electroforesis de DNAElectroforesis de DNA
- 37. ¿Cómo se ve una electroforesis de DNA en geles de Agarosa?
- 38. Electroforesis de DNAElectroforesis de DNA
- 40. 1 2 3 5 4 6 7
- 41. Propiedades estructurales del DNAPropiedades estructurales del DNA 5’p OH3’ 3’OH p5’ Las hebras de una molecula de DNA son Antiparalelas y complementarias
- 42. Complementaridad del DNAComplementaridad del DNA T A C G T A T A C G T A C G C GT A 5’p p 5’ OH3’ 3’ OH Puentes Hidrógeno
- 43. 95°C95°C T G T T C A G CA A C A A G T C GT T A C G T A T A C G T A C G C GT A Desnaturalización/reasociación del DNADesnaturalización/reasociación del DNA
- 44. T G T T C A G CA AC AAG T CG T Reasociación solo con cadenas complementariasReasociación solo con cadenas complementarias
- 45. HibridaciónHibridación Es la unión de dos cadenas de ácidos nucleicosEs la unión de dos cadenas de ácidos nucleicos complementarias para formar un duplex ocomplementarias para formar un duplex o molécula de cadena doble.molécula de cadena doble. • Posibles híbridaciones DNA-DNA DNA-RNA
- 47. Molécula de DNAcs o RNA homóloga a una secuencia de DNAcs o RNA con la que se hibrida de forma estable y específica por asociación de bases complementarias.
- 48. SondasSondas UnaUna sondasonda es un fragmento de DNA que:es un fragmento de DNA que: – Se puede marcar para ser identificado y medidoSe puede marcar para ser identificado y medido – Se hibrida a un DNA gracias a su complementaridadSe hibrida a un DNA gracias a su complementaridad Tipo de marcajesTipo de marcajes – Radioactivo (Radioactivo (3232 P,P, 3535 S,S, 1414 C,C, 33 H)H) – FluorescenteFluorescente – Biotinilación (avidina-streptavidina)Biotinilación (avidina-streptavidina)
- 49. Hibridación de DNAHibridación de DNA inmobilizado en soportes deinmobilizado en soportes de hibridacion:hibridacion: NitrocelulosaNitrocelulosa Membranas de nylonMembranas de nylon Southern BlotSouthern Blot
- 50. NucleasasNucleasas Endonucleasa 5’ Exonucleasa 3’ Exonucleasa
- 51. Southern BlotSouthern Blot Enzimas de retricción DNA de varios tamaños Electroforesis en gel de agarosa gel Denaturación y transferencia a NC blot
- 52. Hibridación de la sonda Visualización
- 53. Southern (and Northern) blot:Southern (and Northern) blot: 1. electrophoresis1. electrophoresis
- 54. Southern (and Northern) blot:Southern (and Northern) blot: 2. transfer to a filter/membrane2. transfer to a filter/membrane
- 56. Southern (and Northern) blot:Southern (and Northern) blot: 3. hybridization to a labeled unique probe3. hybridization to a labeled unique probe
- 59. Testigopositivo Testigonegativo muestra MUESTRA Células, microorganismos autoradiografía hibridacíón lavado sondas Gel de agarosa Ectracción y Denaturalización del ADN Filtro de nitrocelulosa transferencia Separación de filamentos de ADN ELECTROFORESIS
- 60. Southern Blot
- 61. FILTRO DE NITROCELULOSA COLONIAS REPLICA EN PLACA CON FILTRO FILTRO CON COLONIAS COLONIAS ADN DE LAS COLONIAS EN EL FILTRO AUTORADIOGRAFÍA COLONIA - Réplica en placa con filtro - Lisis - Secado - Hibridación con sonda radioactiva
- 62. HibridacionHibridacion in situin situ por fluorescenciapor fluorescencia
- 63. HibridacionHibridacion in situin situ por fluorescenciapor fluorescencia
Notas del editor
- Human Genomic DNA
