Compendio de Biología Molecular Parte 1
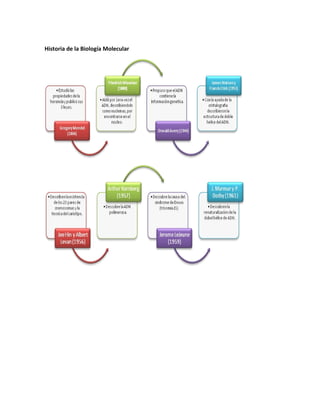
- 1. Historia de la Biología Molecular
- 2. 45 La célula es la unidad mínima del organismo capaz de actuar de manera autónoma. Proyecto del genoma humano Consiste en mapear todos los nucleótidos e identificar los 30000 a 35000 genes presentes en el. Sus objetivos son guardar la información obtenida en bases de datos de libre acceso, desarrollar herramientas para facilitar el análisis de ésta información y trabajar los aspectos éticos, legales y sociales. Algunos de los beneficios son el diagnóstico y prevención de enfermedades, estudio de la susceptibilidad de estas y su tratamiento. Una mutación es el cambio heredable en el material genético de la célula. Tecnología del DNA recombinante 1.- Cortar segmentos del ADN (endonucleasas). 2.- Unir los fragmentos obtenidos (ligasas). 3.- Autoreplicar una molécula de ADN (vectores de clonación). 4.-Insertar los vectores de clonación a células específicas que contienen toda la maquinaria genética para la expresión contenida en el vector. 5.-Seleccionar o identificar a las células que contienen el ADN recombinante. Diferencia entre procariontes y eucariontes Procariontes Eucariontes Organismos Bacterias y cianobacterias Protista, Hongos, Plantas, Animales. Tamaño 1-10 µm 5-100 µm Metabolismo Aerobio Anaerobio Aerobio Organelos Pocos o Ninguno Núcleo, Mitocondria, Cloroplastos, RER y SER.
- 3. 45 ADN Circular y libre Núcleo e Intrones ARN y Proteínas Mismo compartimiento ARN en núcleo y proteínas en citoplasma Citoplasma Sin citoesqueleto Citoesqueleto División Celular Simple Compleja Organización Unicelular Pluricelular con diferentes linajes celulares Canales Organelos Membrana celular • Bicapa lipidica • Colesterol (da fluidez a la membrana) • Glucocaliz (es una capa de alrededor de 50nm que mira hacia el espacio extracelular compuesto principalmente de carbohidratos) • Proteínas integrales • Glucoproteínas Mitocondria • Se formó por un proceso de Endosimbiosis
- 4. 45 • Tiene su propio ADN • Genera el ATP por la cadena de transporte de electrones que se encuentra en la membrana interna • Es de origen materno debido a que las mitocondrias paternas se quedan en la zona intermedia de la cola del espermatozoide • Partes: o Matriz o Membrana mitocondria externa o Membrana mitocondria interna El proceso de integración de la mitocondria a la célula se dio hace millones de años, en el cual, se integró como un parásito y se convirtió en la fábrica de energía de la célula, es por eso que tiene un material genético propio. Lisosomas Degradan componentes, generalmente tienen un pH ácido (5.5), ya que éste ambiente ayuda a las hidrolasas ácidas a degradar todos los componentes que se encuentren dentro de los lisosomas. Existen variantes de estos organelos: • Peroxisomas (ayudan en procesos de detoxificación, eliminando peróxido de hidrogeno) • Proteosomas (es un complejo enzimático que utiliza la vía de la ubiquitinación) Citoesqueleto Ayuda a darle un soporte a la célula, manteniendo su forma y prestándole estabilidad. Se conforma de: • Microfilmentos: Generalmente conformados de actina y miosina. • Filamentos intermedios: Estos varían dependiendo del tejido. • Microtúbulos: Ayudan a transportar vesículas intracelulares además de dar soporte. Núcleo Posee una doble membrana. Podemos encontrar ribosomas unidos a ella. Es importante, ya que es el organelo que protege el material genético de la célula. Algunas de sus partes son: • Nucléolo • Membrana • Poros (un conglomerado de 8 proteínas que ayudan
- 5. 45 al transporte de moléculas que son muy grande para atravesar la membrana por difusión). • Lámina nuclear, conformada con tres proteínas: Laminina A, B y C, que tienen capacidad para fosforilarse o desfosforilarse. Retículo Endoplásmico Se divide en liso y rugoso. A grandes rasgos, las funciones del RER son: síntesis de proteínas y modificaciones post-traduccionales, mientras que en el SER se dan la síntesis de lípidos, esteroides y colesterol, además de procesos de desintoxicación, similares a los que ocurren en los peroxisomas. Una característica importante del SER, es que ayuda a la liberación de calcio que se encuentra recluido en este compartimiento para la contracción muscular. Aparato de Golgi Es morfológicamente similar al RE. Se divide en tres partes: la red cis, la red trans y la red intermedia. Sus funciones son de almacén, síntesis de vesículas y modificaciones de adhesión de carbohidratos complejos. Las vesículas, dependiendo de la señalización celular, podrán migrar a otro organelos o fuera de la célula. El proceso de transporte de biomoleculas puede ser de dos tipos: endocitosis (fagocitosis y pinocitosis) y exocitosis.
- 6. 45 Virus Los virus son complejos macromoleculares, cuyo genoma es un elemento de ácido nucléico el cual se aplica dentro de las células vivas utilizando la maquinaria biosintética de éstas para dirigir la síntesis de partículas especializadas, los viriones; los cuáles contienen el genoma viral y lo transfieren a otras células. No es un ser vivo, es un ente ya que posee material genético y de esa manera podríamos dividirlos en: Retrovirus (RNA) y Adenovirus (DNA). Además de poder ser bicatenoides o monocatenoides. Por la forma de la cápside en: Cilíndricos, helicoidales, icosaedricos, complejos. También pueden ser divididos en: vegetales, animales y bacteriófagos, dependiendo del tipo de células a la que parasitan. Si tienen cubierta lipoprotéica, puede dividirse en envueltos o desnudos. Proceso de infección 1.-Fijación o adsorción: Este proceso inicial de la infección consiste en la unión de ciertas proteínas específicas, que principalmente se hallan en la cápside de los virus encapsulados, a receptores de membrana que medían la endocitosis de partículas, para que sean introducidos al citoplasma. Los virus no encapsulados, pueden en algunos casos entrar por difusión simple. 2.- Penetración: Con la ayuda de los receptores de membrana, los virus pueden entrar envueltos en vesículas recubiertas con clatrina, o sin envoltura. 3.- Decapsidación: Si se trata de virus encapsulados ocurre un proceso de remoción de la cubierta del material. Puede estar dada por enzimas propias del virus, o bien puede ocurrir una fusión entre el endosoma que recubre al invasor, y un proteosomas para que así las proteasas propias de la células digieran la cápside viral, y así queda libre el material genético para su posterior uso.
- 7. 45 4.-Replicación del material genético: Sucede de forma similar que el material propio, en la mayoría de los casos. 5.- Expresión o Integración: Se integra el material viral al material celular para que suceda la transcripción del genoma de manera simultánea. 6.- Ensamblaje: Procesamiento de ARNm viral a proteínas. 7.- Gemación: Expulsión del virus “hijo”. Priones Se cree que los priones se diferencian de los virus, porque pueden consistir en proteínas y no ácidos nucléicos. Teóricamente las proteínas de los priones son codificadas por genes celulares y actúan como señales reguladoras. Hay evidencias que sugieren que ciertas enfermedades neurodegenerativas como el Kuru, pueden ser causadas por priones.
- 8. 45 Manipulación de macromoléculas. Clasificador de moléculas activado por fluorescencia. Se determina la fluorescencia de las células que pasan a través de un rayo laser. Las gotitas que contienen una sola célula reciben una carga negativa o positiva según la fluorescencia. Posteriormente las gotitas son desviadas en función de su carga mediante un campo eléctrico hacia los tubos de recogida. Microdisección por fractura láser Utiliza un rayo laser para escindir una región de interés y capturarla a un contenedor. Placa de cultivo Los cultivos se preparan a partir del mismo organismo (primarios) o de otro cultivo (secundarios). Necesitan de una superficie para poder crecer, un pH fisiológico y componentes de la matriz extracelular como colágena o laminina. Heterocariontes Es el resultado de la fusión de una célula con otra formando una célula combinada con 2 núcleos independientes. Posteriormente darán lugar a hibridomas.
- 9. 45 Anticuerpos monoclonales Se basa en propagar un clon de células a partir de un solo linfocito B secretor de anticuerpos, pero estos tienen una vida limitada en cultivo, por lo cual se fusionan con células derivadas de un tumor inmortal de linfocito B. Estos hibridomas se propagan como clones, cada uno de los cuales constituye una fuente permanente y estable de anticuerpo monoclonal. Centrifugación Los preparados de células se exponen a altas velocidades de giro y se separan en
- 10. 45 función de su tamaño y densidad dejando sobrenadante (líquido) y un pellet (remanente de estructuras). Cromatografía Técnica en la que una mezcla de proteínas en solución se hace pasar a través de una columna que contiene una matriz sólida porosa. Las diferentes proteínas de la mezcla se van retrasando a causa de su interacción con la matriz. En función del tipo de matriz que se utilice las proteínas se pueden separar: • En función de su carga (Cromatografía de intercambio iónico) • De su hidrofobicidad (Cromatografía hidrofóbica) • De su tamaño (Cromatografía de filtración) • De su capacidad para unirse a determinados grupos químicos (Cromatografía de afinidad). • Existe otro tipo llamada HPLC (Líquida de Alta Resolución), en la cual nuevas resinas cromatográficas basadas en sílice en forma de diminutas esferas ayudan al proceso y se alcanza un alto grado de separación. Electroforesis Las proteínas tienen carga. Si se aplica un campo eléctrico a una solución que contenga una molécula proteica, esta migrara a una velocidad que dependerá de su carga neta, de su tamaño y de su forma.
- 11. 45 • SDS-Page (Electroforesis en gel de poliacrilamida con detergente duodecil sulfato sódico): Se usa como matriz inerte un gel de poliacrilamida y el detergente cargado negativamente SDS. Además se suele añadir un agente reductor como el β-mercaptoetanol para romper enlaces disulfuro. Cada molécula de proteína se une a gran cantidad de moléculas del detergente, cargadas negativamente, que enmascaran la carga intrínseca de la proteína y hacen que cuando se aplica un voltaje la proteína migre hacia el electrodo positivo. • Electroforesis bidimensional: Combina 2 procedimientos diferentes. En el primer paso las proteínas se separan en función de su carga. Luego las cadenas polipeptídicas se fraccionan mediante un procedimiento denominado isoelectroenfoque, que se basa en que la carga neta de una proteína varia con el pH de la solución. Confección de un mapa peptídico o huella dactilar de una proteína La proteína es digerida por enzimas (tripsina) para generar una muestra de fragmentos, que luego se fraccionan en 2 dimensiones mediante electroforesis y cromatografía de partición. Esta última técnica separa a los péptidos en función de su solubilidad en agua, que se une preferentemente a la matriz sólida, y en el solvente en el que son aplicados. El patrón de manchas obtenido, tiene valor diagnóstico para la proteína analizada y también se usa para detectar modificaciones postraduccionales de las proteínas. Espectrometría de masas
- 12. 45 Puede usarse para secuenciar fragmentos peptídicos e identificar proteínas. Nos permite determinar la masa exacta de proteínas intactas o de péptido derivados de ellas por ruptura enzimática o química. Cristalografía de rayos X Es la principal técnica que se ha usado para determinar la estructura tridimensional. Si se dirige un haz estrecho y paralelo de rayos X a una muestra de una proteína pura la mayoría pasaran a su través. Sin embargo una pequeña fracción serán dispersados por los átomos. Si ésta es un cristal bien ordenado los haces dispersados se reforzaran unos a otros en cierto puntos y aparecerán como manchas de difracción cuando sea recogidas en un detector. Experimento de Hershey y Chase Se utilizaron bacteriófagos en este experimento además de elementos radioactivos como 32P y 35S. Si la información genética estaba contenida en el ADN la marca radiactiva debía estar en el interior de las bacterias de este último grupo, por el contrario si eran las proteínas las que cumplían dicha función la marca radiactiva estaría adentro de las bacterias del primer grupo. El resultado del experimento confirmó que el ADN era la molécula que buscaban, ya que se encontraba la marca radioactiva en el interior de las bacterias que se pusieron en contacto con ADN marcado. Experimento de Avery
- 13. 45 En éste experimento se demostró que al añadir ADN purificado a una bacteria cambian sus características y que los cambios pasan a las siguientes generaciones. Se utilizaron 2 sepas próximas de la bacteria Estreptococus Pneumoniae. Una sepa tiene aspecto liso (S) y cuando se inyecta en ratones les provoca la muerte; la otra parece rugosa (R) y es no letal. Los resultados muestran que una sustancia presente en la sepa S puede cambiar o transformar la sepa R en la sepa S y que éste cambio es heredable. Estructura del material genético: ADN y ARN ADN Polinucleótido formado por la unión covalente entre unidades de desoxirribonucleótidos, actúa como transportador de la información genética. Los monómeros o nucleótidos que forman las cadenas de ADN tienen la siguiente estructura: • Un grupo fosfato • Un azúcar, desoxirribosa. • Una base nitrogenada ya sea una purina o pirimidina. Las bases nitrogenadas son:
- 14. 45 Postulados de Chargaff • El número de Adeninas es igual al número de Timinas. • El número de Guaninas es igual al de Citocinas. • Las proporciones de A-T y G-C son casi las mismas en todas las especies. Modelo de Watson y Crick 1. Existen 2 cadenas helicoidales de polinucleótidos enrolladas a lo largo de un eje en común. Las cadenas transcurren en direcciones opuestas (son antiparalelas). 2. Los ejes azúcar-fosfato se sitúan en el exterior y por tanto las bases en el interior. 3. Las bases son casi perpendiculares al eje de la hélice y las bases adyacentes están separadas 3.4 Å. La estructura helicoidal se repite cada 34 Å, de modo que hay 10 bases por cada vuelta de hélice. Hay una rotación de 36° por base. 4. El diámetro de la hélice es de 20 Å. La interacción que mantiene unidas a las 2 cadenas son puentes de hidrogeno (interacciones débiles entre las bases) que se aparean de forma complementaria: • 2 puentes de Hidrógeno en la unión de Timina con Adenina • 3 puentes de Hidrógeno en la unión de Citosina con Guanina Además los polinucleótidos se leen del extremo 5´al 3´y la orientación de las 2 cadenas es antiparalela. Por último, el ADN visto a groso modo y de una manera un tanto macroscópica presentará surcos en su periferia de tal modo que se dividirán en surco mayor y surco menor.
- 15. 45 ARN Tiene características muy similares al ADN a excepción de que su azúcar es una ribosa y no una desoxirribosa y además no utiliza la base Timina (T), sino Uracilo (U). Se encuentra principalmente en una sola hebra y no en una doble hélice, ya que pierde su estabilidad al tener un OH en la posición 2 en vez de un H, como en el caso del ADN. Existen tres tipos principales de ARN: • mARN: Transporta las instrucciones del ADN a los ribosomas para formar proteínas. Se obtiene a partir de un templado de ADN mediante la transcripción. • rARN: Forma parte de los ribosomas, que se involucran en la traducción de RNA a proteínas. Es el tipo más común en las células representando alrededor del 50% • tARN: Tiene la función del traductor, pues cambia el mensaje del mARN a nucleótidos, para la síntesis de proteínas. Tiene una conformación diferente ya que se encuentra en una forma plegada y consta de 3 asas. Está formado por 73 a 90 nucleótidos. Encontramos en su extremo 3´el aminoácido y en la segunda asa el anticodón. Este tipo de ARN habla los 2 idiomas (nucleótido y aminoácido) Detección de nucleótidos Las bases apiladas de los ácidos nucléicos absorben menos luz UV que las bases que no lo están, efecto que se
- 16. 45 conoce como hipocromismo. Por tanto, la fusión de ácidos nucléicos se puede seguir fácilmente midiendo su absorbancia de luz que tiene un pico de una longitud de onda de 260 nm. Existen en la naturaleza 3 tipos de ADN: (la forma B es la mas común) A B Z Sentido de la hélice Dextrógiro Dextrógiro Levógiro Diámetro 26 Å 20 Å 18 Å Pares de bases por vuelta 11 10.5 12 Altura de la vuelta por par de bases 2.6 Å 3.4 Å 3.7 Å Separación entre las bases y el eje de la hélice 20° 6° 7° Conformación de las azucares C-3´ C-2´ C-2´ para pirimidinas; C-3´ para purinas Conformación de enlace glicosílico Anti Anti Anti para pirimidinas Syn para purinas Propiedades del ADN 1. Desnaturalización: es el proceso en el que la doble cadena de ADN se separa, a menudo altas temperaturas. a. Tm temperatura de fusión, se define como aquella temperatura en la cual se deshace la mitad de la estructura helicoidal. 2. Renaturalización por complementariedad: es el proceso por el cual las 2 cadenas separas de ADN se unen de nuevo buscando su base complementaria. 3. Hibridación: es el proceso por el cual una cadena de una ADN ajeno o extraño, se une a otra cadena siguiendo el mismo principio de complementariedad.
- 17. 45 Secuencia palindromica Secuencia de nucleótidos que es idéntica a su cadena complementaria cuando cada una es leída en la misma dirección química. Repetición en espejo 2 copias de una misma secuencia orientadas en sentido contrario en una misma hebra Cromosomas Un cromosoma es la estructura compacta y ordenada que resulta del plegamiento del ADN, son portadores físicos de los genes. Están formados por una molécula única de ADN lineal muy larga que contiene varios genes. Un ser humano está constituido por 46 cromosomas (22 pares de autosomas y 1 par de sexuales X,Y).
- 18. 45 Cariotipo Es el estudio sistemático de los cromosomas. Formación de un cromosoma Los elementos incluidos en la formación de un cromosoma son: • Cromatina: Complejo de ADN y proteínas (heterocromatina y eucromatina) • Proteínas especializadas: Histonas y no histonas • Nucleosomas: complejo de 8 histonas (octámero) y proteínas especializadas en los que se enrolla una hebra de ADN. Nucleosoma Es una estructura que constituye la unidad fundamental y esencial de cromatina, que es la forma de organización del ADN en las eucariotas. Los nucleosomas están formados por un núcleo constituido por un octámero de histonas. El octámero está formado por dos moléculas de cada una de las histonas H2a, H2b, H3 y H4. Viéndolo en
- 19. 45 un microscopio electrónico, se ve con forma de rosario o "collar de perlas", ya que está formada por la doble hélice de ADN enrollada sobre sucesivos octámeros de histonas, existiendo entre dos nucleosomas consecutivos un fragmento de ADN, ADN espaciador. Cada octámero de histonas está rodeado por casi 2 vueltas de ADN bicatenario. Otro tipo de histona (H1) se extiende sobre la molécula de ADN fuera de la parte central del nucleosoma. Un nucleosoma se encuentra cada 200 pares de bases. Replicación del ADN Proceso mediante el que se produce una copia idéntica del material genético de una célula. Este proceso sucede principalmente cuando va a suceder una división celular (mitosis y meiosis). En cada ciclo de replicación se utiliza cada una de las 2 cadenas de ADN como patrón para la formación de una cadena complementaria de ADN. De este modo, las cadenas originales se mantienen intactas a lo largo de muchas generaciones celulares. A este mecanismo se le llamó replicación semi conservativa.
- 20. 45 Proteínas involucradas en la replicación • Proteínas iniciadoras: Reconocen el origen de la replicación • Helicasas: Son proteínas que desenrollan la doble hélice rompiendo los puentes de H que unen a las cadenas. • Proteínas que separan a las cadenas: o Proteínas desestabilizadoras: generan una región recta en el ADN. o Girasas: liberan parte de la presión por giros del ADN. o SSB (Single-Strand Binding Protein): Se unen a cadenas sencillas de ADN para estabilizar la separación. • ADN polimerasa: une nucleótidos complementarios. • ADN ligasa: Une fragmentos de Okazaki. • ADN Topoisomerasa: Evita el enrollamiento del ADN, quitando presión. • Primosoma: Complejo formado por la helicasa y la primasa (sintetiza primers o cebadores). Horquilla de replicación Es una región concreta de replicación que se desplaza a lo largo de la doble hélice paterna de ADN. Tiene una estructura en forma de Y (de ahí su nombre). En esta horquilla se sintetiza el ADN de las 2 hélices hijas mediante un complejo multienzimático que contiene ADN polimerasa. (Esta enzima sólo sintetiza una nueva cadena de dirección 5´a 3´) Una horquilla de replicación tiene una estructura asimétrica. La cadena hija de ADN que se sintetiza de manera continua se llama cadena conductora, es sintetizada antes de la otra cadena hija que crece de forma discontinua y que recibe el nombre de cadena retrasada. La síntesis de la cadena retrasada es más lenta debido a que ha de esperarse a que la cadena conductora exponga la cadena patrón sobre la que se sintetizará cada uno de los fragmentos de Okazaki Un fragmento de Okazaki es una zona en el ADN de la horquilla de crecimiento que se sintetizan en una dirección de 5´a 3´ y que después de su síntesis se unen a las cadenas de ADN mediante las ADN ligasas. Mecanismos de corrección
- 21. 45 1. Corrección de galeradas: tiene lugar justo antes de la adhesión de un nuevo nucleótido a la cadena en crecimiento. El nucleótido correcto tiene mayor afinidad por unirse a su base complementaria. 2. Corrección de galeradas exonucleótidas: en donde el domino exonucleasa de la ADN polimerasa puede eliminar un nucleótido ya apareado (tautomeros), para corregir. De esta manera la ADN polimerasa actúa como una enzima autocorrectora. Complejo Primosoma (Primasa y Helicasa) Para el caso de la cadena conductora únicamente es necesario un cebador especial al inicio de la replicación: en cuanto se ha establecido la horquilla de replicación, la ADN polimerasa dispone de un extremo de cadena con bases apareadas sobre el que sintetizar la nueva cadena. Sin embargo en la parte de la cena retrasada, cada vez que la ADN polimerasa concreta un corto fragmento de ADN (fragmento de Okazaki), ha de empezar a sintetizar otro fragmento completamente nuevo en un punto más delante de la cadena patrón. Es entonces donde entra la ADN primasa que es la enzima que sintetiza los cortos cebadores de ARN sobre la cadena retrasa utilizando ADN como patrón. A diferencia de la polimerasa, esta enzima puede iniciar una nueva cadena de polinucleotidos uniendo entre si 2 nucleosidos trifosfato. La primasa sintetiza un Polinucleótido corto en dirección 5 ´a 3´y luego se detiene, dejando el extremo 3´ de este cebador accesible a la ADN polimerasa ADN Helicasa Esta enzima se desplaza rápidamente por las cadenas de ADN. Cuando encuentran una región de doble hélice continúan desplazándose por la misma cadena, desenrollando así la hélice para que de esta manera la ADN polimerasa y la ADN primasa puedan hacer su trabajo. Básicamente su mecanismo de acción es romper los puentes de hidrógeno entre las bases apareadas utilizando ATP como fuente de energía. Dicha enzima consta de 6 subunidades y tiene 4 sitios que hidrolizan ATP.
- 22. 45 Proteínas de unión al ADN o SSB También son llamadas proteínas desestabilizadoras de las hélices (Single-Strand Binding Protein). Se unen fuertemente y de un modo cooperativo a las cadenas de ADN abiertas, sin tapar las bases de la cadena que quedarán entonces disponibles para actuar como un patrón. Colaboran con las Helicasas estabilizando la conformación desenrollada de cadenas sencillas. Constan de 2 dominios: A y B Acarreador de la ADN polimerasa Este acarreador también llamado abrazadera mantiene a la polimerasa firmemente unida al ADN cuando se desplaza, pero la libera en cuanto la polimerasa se detiene ante una región de doble cadena. La proteína abrazadera forma un amplio anillo alrededor de la hélice del ADN. Un lado se une a la parte trasera de la ADN polimerasa y el anillo completo se desliza libremente a medida que la polimerasa se desplaza por la cadena de ADN. El ensamblaje de la abrazadera requiere de hidrólisis de ATP, mediante un complejo de proteínas especial, el cargador de la abrazadera al patrón cebador. El cargador recibe el nombre de Clamp loader y la abrazadera de sliding.
- 23. 45 Complejo Mut También llamado el scanner del ADN, recorre la cadena y en cuanto ve a un apareamiento no perfecto lo corta y después lo repara evitando así errores en la replicación. Existen 2 tipos: • Mut L: busca una muesca (el resultado del error de apareamiento) • Mut S: se une al par de bases mal apareado Súper enrollamiento A medida que una horquilla de replicación se va desplazando a lo largo de una doble cadena de ADN genera problemas de enrollamiento. Es por eso que durante la replicación se utilizan topoisomerasas que forman una plataforma giratoria en la hélice del ADN. Las topoisomeras son de 2 tipos: • I .- rompe enlaces fosfodiester y no permite en super enrrollamiento • II.- Impide que se vuelva a formar la doble hélice y no necesita ATP
- 24. 45 Orígenes de replicación Son las posiciones por las que se abre en primer lugar la hélice de ADN, este ADN contiene secuencias cortas que atraen proteínas iniciadoras. Estas secuencias suelen ser ricas en Adenina y Timina por lo que es más fácil separarlas debido a que solo existe una interacción de 2 puentes de hidrógeno en esta unión. En células eucariotas existen varios sitios de replicación, mientras que en las bacterias solo existe uno (Ori C) Replicón Es la unidad de ADN en donde ocurre un evento de replicación. Cada replicón inicia solamente durante un ciclo celular y tiene elementos de control en el origen y la terminación. Sus funciones son: 1. Iniciar el ciclo de replicación 2. Controlar la frecuencia de eventos 3. Segregar los cromosomas replicados Adición de nuevas histonas al ADN por detrás de la horquilla de replicación. Las histonas nuevas sufren acetilación, durante la fase S (síntesis) del ciclo celular. Los telómeros son el sitio final de los cromosomas. Con cada ciclo celular y por consiguiente por cada replicación dicho espacio se va a acortando. Es entonces en donde una enzima llamada Telomerasa que posee 2 subunidades (un molde de ARN, y un sitio catalítico que a partir del molde sintetiza cadenas complementarias que solo pueden unir secciones de G y T) evita el acortamiento de los telómeros. Está científicamente demostrado que este proceso de acortamiento de los cromosomas contribuye al envejecimiento, por ello se han hecho experimentos utilizando una telomerasa exógena en ratones para tratar de hacer mas larga la vida de estos, sin embargo, el efecto en los resultado ha sido la generación de neoplasias.
- 25. 45 Transcripción Es el proceso mediante el cual se obtiene ARN a partir de un molde de una molécula de ADN. Éste proceso sucede en el núcleo en las células eucariotas, mientras que en las procariotas sucede en el citoplasma. En las células eucariontes, el templado (molde) de ADN codifica para un solo gen, por lo que se le denomina monocistrónico, mientras que en los procariontes, en la misma secuencia existen varios genes diferentes, a lo que se le llama policistrónico. Los elementos necesarios para que éste proceso se lleve a cabo son: • Región promotora en la cadena molde (se explica más adelante): sirve para el acoplamiento de la ARN polimerasa. • ARN polimerasa II: Ensambla la cadena de RNA, necesita de una fosforilación para “activarse”. • Complejo de inicio • Ribonucleótidos • Plantilla molde de ADN. Transcripción en procariotas Tienen un solo tipo de ARN polimerasa, utilizan el factor Sigma el cual aumenta la afinidad de la polimerasa por los sitios promotores; una vez iniciando la transcripción se separa. Etapas de la transcripción 1. Se asocia la polimerasa con la plantilla de DNA. 2. La polimerasa de desplaza en dirección 3´a 5´ a lo largo de la cadena molde de ADN ensamblando una cadena complementaria antiparalela de ARN que crece en dirección 5´a 3´. 3. Una vez que la polimerasa pasa sobre un sitio en particular se reconstruye la doble hélice, por lo que no es necesario utilizar helicasas como en la replicación. 4. Los nucleótidos que ensambla la polimerasa en la cadena de RNA naciente, son complementarios a la cadena molde de DNA. 5. La molécula naciente se denomina mARN.
- 26. 45 Maduración del mRNA Para que el mRNA pueda salir del núcleo al citoplasma, se necesita que éste madure. Esto se da gracias a ciertas modificaciones postranscripcionales que ocurren dentro del núcleo. Son las siguientes: 1. Corte y empalme: Dentro de la molécula de RNA mensajero existen regiones que no codifican para proteínas, por lo que deben ser removidas del mRNA. Estas regiones se conocen como Intrones (80-10,000 nucleótidos). Las regiones sobrantes que si tienen un papel en la síntesis de proteínas, se conocen como exones. Para que los intrones sean removidos sucede lo siguiente: o Dentro de los intrones existen regiones “conservadas” que ayudan a que esta secuencia sea removida de la cadena. Estas regiones son GU, A, y AG. o La región GU (que generalmente se encuentra cercana a un exón anterior), comienza a unirse a la A (que generalmente se encuentra en medio de las otras dos regiones), formando así un lazo (lariet) en la cadena. Esta unión sucede por una transesterificación. El sitio A es reconocido por una proteína BBP. o La parte 5’ del lazo, es cortada por un complejo de proteínas (U), que se denomina splisosoma. Éste complejo también corta la región AG, que aún está unida al exón siguiente, mediante una segunda transesterificación. o Finalmente los extremos de los exones son unidos. o En ciertos casos este proceso no ocurre de forma correcta por lo que se aumenta o disminuye el tamaño de los exones creando lo que se conoce como Sitios de Corte Críptico, que son sitios de corte alternativo. Enfermedades como las talasemias (defectos en la síntesis de hemoglobina) son causadas por estos sitios alternos.
- 27. 45 2. Metilación: Para mantener la estabilidad de la cadena de mRNA, es necesario que se añada un “capuchón” en el extremo 5’ de la cadena. Éste capuchón es una metilación (7-metilguanosina) que se añade a la cadena. 3. Poliadenilación: Se codifica una señal en el lugar donde terminó la transcripción (sitio 3´de la cadena). Una enzima llamada Polimerasa de Poli-A (PAP), comienza a añadir adeninas al final de la cadena creando lo que se conoce como una cola de Poli-A. También se añaden ciertas proteínas que cubren la cola de Poli-A. Entre mayor sea el número de Adeninas mayor estabilidad tiene la cadena. Los factores CAP cubren la poliadenilación para proteger la cadena. 4. Factores de exportación: Proteínas como el CBC (que cubre la metilación), SR (que cubren los sitios UTR), y otros factores, son necesarios para que el mRNA pueda salir del núcleo para ser traducido en el citoplasma. Muchos de estos factores se quedan en el núcleo al momento que la cadena atraviesa el poro por donde pasa al citoplasma.
- 28. 45 Traducción La traducción es el proceso por el cual, una hebra de mRNA, se traduce de idioma “nucleótido” a idioma “aminoácido”, para la creación de una proteína, dentro del RER. Ésta traducción se realiza en base a la secuencia de nucleótidos, que se agrupan en grupos de 3 (tripletes), llamados codones. Cada codón codifica para un aminoácido o para que se detenga la traducción. La secuencia de codones que codifican para los diferentes aminoácidos se denomina código genético. Existen diferentes elementos involucrados en la traducción: • mRNA • tRNA, que es el tipo de RNA, que traduce el mensaje escrito en el mensajero. • Ribosomas, en los cuales sucede la traducción y el ensamblaje de las proteínas. • Factores que regulan la traducción. mRNA Se trata de una molécula de RNA mensajero ya maduro (sin intrones y con modificaciones postranscrpcionales), que salió del núcleo para ser traducida en el RER. Consta del factor eIF (factor de inición de la traducción)
- 29. 45 tRNA Es el RNA que se encarga de traducir el mensaje codificado en el mRNA. Un ARNt está formado por entre 73 y 90 nucleótidos. Se encuentra disuelto en el citoplasma celular. Pueden presentar nucleótidos poco usuales como ácido pseudouridílico, ácido inosílico e incluso bases características del ADN como la timina. Este ARNt es procesado por la ARN polimerasa III, y al igual que los demás ARN, necesita de un proceso de maduración antes de salir del núcleo y dirigirse hacia el citoplasma. El ARNt presenta zonas de complementariedad intracatenaria, es decir, zonas complementarias dentro de la misma cadena, lo que produce que se apareen dando una estructura característica semejante a la de un trébol de tres hojas. Tiene las siguientes características: • Brazo aceptor formado por el extremo 5' y el extremo 3', que en todos los ARNt posee la secuencia CCA, cuyo grupo -OH terminal sirve de lugar de unión con el aminoácido. • El bucle TΨC, que actúa como lugar de reconocimiento del ribosoma. • El bucle D, cuya secuencia es reconocida de manera específica por una de las veinte enzimas, llamadas aminoacil-ARNt sintetasas, encargadas de unir cada aminoácido con su correspondiente molécula de ARNt. Dichas enzimas constan de 2 mecanismos para agregar el aminoácido correcto y de esa manera evitar errores en la traducción. • El bucle situado en el extremo del brazo largo, que contiene una secuencia de tres bases llamada anticodón. Cada ARNt "cargado" con su correspondiente aminoácido se une al ARNm, mediante la región del anticodón, con tripletes de bases del ARNm (cada tres bases del ARNm definen un triplete o codón) en el proceso de la traducción de la información genética que conduce a la síntesis de las proteínas. Los aminoácidos son añadidos al extremo C-terminal de la cadena polipeptídica en crecimiento generando enlaces peptídicos en donde cada aminoácido añadido aporta la energía necesaria para su unión y también la adhesión del siguiente aminoácido
- 30. 45 Ribosomas Son estructuras que se encuentran en el RER, encargadas de la síntesis de proteínas. Se componen de rRNA y de proteínas. Se dividen en 2 subunidades con diferentes coeficientes de sedimentación. La subunidad menor contribuye con el soporte sobre el que los ARNt pueden aparearse correctamente con los codones del ARNm y la subunidad mayor cataliza la formación de los enlaces peptídicos uniendo los aminoácidos para conformar una proteína. Cuando no están fabricando dicha proteína, las 2 subunidades se encuentran disociadas. Cuando se encuentra un codón de paro (UAG, UGA, UAA), el ribosoma libera la proteína terminada y sus 2 subunidades se disocian de nuevo, con lo que pueden ser reutilizadas para sintetizar otra proteína. Los ribosomas procariotas se denominan 70S (subunidades de 50S y 30S) y los eucariotas 80S (subunidades de 60S y 40S). Tienen 3 sitios importantes para la traducción, el sitio A (anclaje), P (polipéptido) y E (de exit o salida).
- 31. 45 Mecanismo de la traducción: 1. Iniciación 1.1. El ARN maduro es reconocido por factores de iniciación. 1.2. Unión del iniciador ARNtMet (iniciador) a la subunidad menor del ribosoma 1.3. Unión de este complejo de ARNm y localización del codón de inicio (AUG) 1.4. Unión de la subunidad mayor del ribosoma *Factores de iniciación eIF formador por: • Metionina • ARNt • eIF2 • GTP 2. Elongación 2.1. Un ARNt que transporta el próximo aminoácido que se debe incorporar a la proteína se une al centro A mediante el apareamiento de su anticodón con el codón del ARNm situado frente de él, de manera que tanto el centro P como el centro A tienen unido ARNt 2.2. El extremo carboxilo de la cadena se desprende de su ARNt en el centro P y se une al grupo amino libre del aminoácido unido al ARNt del centro A formándose un nuevo enlace peptídico (reacción catalizada por la peptidil transferasa contenida en la subunidad mayor del ribosoma) 2.3. Otra serie de cambios conformacionales hace que el ARNm se desplace una distancia equivalente exactamente a 3 nucleótidos a lo largo del ribosoma y deja a este preparado para recibir al nuevo aminoacil-ARNt recién llegado. El paso se repite varias veces hasta encontrar un codón de termino. *Factores de elongación (hidrolizan GTP a GDP) • EF-TU • EF-G 3. Terminación 3.1. Finaliza el proceso cuando aparece un codón de termino (UAA, UAG y UGA) 3.2. Los factores de liberación se unen a cualquier ribosoma que tenga un codón de paro en el sitio A, y esta unión fuerza a la peptidil transferasa a catalizar la adhesión de una molécula de agua al peptidil ARNt en lugar de un aminoácido. 3.3. Entonces se libera el extremo carboxilo terminal de la cadena polipeptídica en crecimiento
- 32. 45 3.4. El ribosoma libera al ARNm y se seperan las subunidades para ser reutilizadas posteriormente. Traducción Procariota Eucariota Ribosoma 30S y 50S 40S y 60S ARNm Policistrónico Monocistrónico Unión al ribosoma Shine-Delgarno y AUG CAP 5´y AUG Regulación de la transcripción Si ¿? Iniciación IF1, IF2, IF3 eIF 1, 2, 3, 3, 5 Elongación RF-1, RF-2, RF-3 eRF1 y eRF3 Terminación EF-Tu, EF-Ts, EF-G eF1a, eF1b y EF2 Formación del complejo de inicio En procariotas: • IF2 e IF1 se unen al sitio P de la subunidad mayor (50S) • La subunidad pequeña se asocia con el ARNm, en donde IF1 se une al sitio A • IF2 con GTP ayuda a que el ARNt se une al complejo y posteriormente se disocian los IF En eucariontes: • eIF4F: une al extremo 5´ y forma el complejo Ternario eIF4E, eIF4A, eIF4B • eIF4E: es un punto de regulación • eIF3: ayuda a que la subunidad pequeña del ribosoma interactúe con eIF4G que interactua con eIG4F y de esa manera el ribosoma reconoce al ARNm. Este factor eIF3 se encuentra en la subunidad pequeña y el eIF6 en la subunidad grande, ambos son necesarios para mantener a sus respectivas subunidades en estado disociado y se liberan cuando la subunidad mayor se une al complejo de inicio
- 33. 45 Polirribosoma o Polisoma Son largos ensamblajes citoplasmáticos formados por varios ribosomas separados entre sí únicamente por 80 nucleótidos, situados a lo largo de la misma molécula de ARNm. Permiten que se puedan fabricar muchas mas moléculas de proteínas en un tiempo determinado. Control de Calidad Existe la posibilidad de que el ARNm listo para codificarse a el lenguaje aminoácido sea incorrecto o presente errores ya que, aunque exista un buen proceso de maduración en el nucleo, el ARNm puede romperse en el citosol. Es por eso que el complejo de inicio verifica en una primera instancia que exista el capuchón 5´y la cola poli A 3 ´antes de empezar con el proceso de traducción. Las bacterias (procariontes) solucionan el problema de los ARNm incompletos de un modo completamente diferente. No solo hay señales en los extremos 3´de los ARNm bacterianos, si no que además la traducción frecuentemente empieza antes de que se haya completado la síntesis del transcrito. Cuando el ribosoma bacteriana traduce un ARNm incompleto hasta el final, el sitio A del ribosoma entra un ARN especial (ARNtm ARN templado), que es traducido a continuación del ARNm truncado; esto añade una señal de 11 aminoácidos (alaninas) al extremo carboxilo terminal de la proteína truncada que indica a las proteasas que toda la proteína tiene que ser degradada.
- 34. 45 Codones de paro tempranos Existen variaciones poco comunes en el código genético La selenocisteína es esencial para el correcto funcionamiento de una gran variedad de enzimas. Se produce a partir de una serina unida a una molécula especial de ARNt que se aparea con el codón UGA (codón de paro). Los ARNm de las proteínas en las que se ha de insertar la selenocisteína en un codón UGA tienen una secuencia especial de nucleótidos (en forma de horquilla) próxima a ese codón que provoca esta recodificación. Caminos de las proteínas • Se pueden plegar, este plegamiento puede darse por: o Sin ayuda o Con chaperonas o Con energía o Con un complejo enzimático • Pueden sufrir modificaciones post traduccionales • Incorporación de subunidades para formar una estructura cuaternaria (por ejempolo la Hemoglobina) Si no existe plegamiento de la proteína hay degradación en aminoácidos a menudo porque la proteína no se encuentra de un modo correcto o porque proviene de un ARN mensajero truncoal cual ha sido añadido un ARNtm (templado).
- 35. 45 Chaperoninas Las proteínas mal plegadas son captadas mediante interacciones hidrofóbicas con uno de los márgenes del barril. La unión de ATP y de una proteína que actúa como cubierta aumenta el diámetro del barril, que puede forzar la entrada de la proteína diana parcialmente plegada. Entonces la proteína queda confinada en un espacio cerrado, donde tiene una nueva oportunidad de plegarse completamente. Tras unos 15 segundos la hidrólisis del ATP expulsa la proteína y el ciclo vuelve a comenzar Proteosoma Abundante proteasa dependiente de ATP que constituye casi el 1% de la proteína celular. Existen muchas copias dispersas por el citosol y por el núcleo, también actúa sobre proteínas del RER: las que no se pliegan o no se ensamblan bien siguen la vía de la Ubiquitinación y finalmente son destruidas. Genes Es un ácido nucleico (ADN y ARN) que codifica para una proteína (enzima o proteína estructural) o un ARN (ARNt; ARNr, ARNnp, ARNm) incluyendo sus secuencias de regulación.
- 36. 45 Es un segmento corto de ADN que codifica para una proteína en específica y que se encuentra en los cromosomas, ocupando un lugar en específico denominado LOCUS. Este es un factor que transmite la herencia. El término de gen fue denominado por Gregorio Mendel el cual por medio de sus leyes y sus observaciones en las semillas (verdes y amarillas), descubrió lo siguiente: 1° LEY: Al aparear las dos razas puras sus descendencias serán iguales. 2° LEY: Mendel tomo la primera descendencia del apareamiento de la raza pura (F1), en la cual observo que aunque este no exprese alguna característica que es recesiva, esta la puede transmitir a su descendencia y estas las pueden expresar. 3°LEY: En la consecuencia con lo expresado en la 2° ley se observo que los rasgos heredados son independientes unos de otros, por lo tanto un patrón de herencia no es dependiente de otro. Clasificacion de los genes: Los alelos por lo tanto se definen como una de las formas alternativas en las cuales se puede expresar un gen, puede ser: • Dominantes: son aquellos que se expresan. • Recesivos: son aquellos que no se expresan en el fenotipo pero que sin embargo se expresan cuando hay dos alelos recesivos. Otro tipo de clasificación es la siguiente: • Codificantes o estructurales: se encuentran expresados en todas las células de un organismo • Reguladores: cuya función es determinar el tejido, momento y cantidad de la proteína, es decir, le dan especificidad a la célula. Gen estructural que codifica una proteína implicada en la regulación de la expresión de otros genes. Codifica una proteína que controla transcripción mediante la unión a un sitio o sotios particulares del ADN: Cis: secuencia de ADN que no es convertida en otra forma, sino que funciona solamente como una secuencia in situ, afectando solamente al ADN al que está ligado. Trans: secuencia de ADN que codifica una proteína libres de difundir para llegar a su diana. Los genes están localizados en los cromosomas en el núcleo celular y se disponen en línea a lo largo de cada uno de los cromosomas. Un Locus es la región en donde se localiza un gen Loci son los sitios donde se localizan varios genes en un mismo cromosoma
- 37. 45 proteínas de acción trans: factores de transcripción que se ynen a secuencias de DNA reguladoras de acción cis. Cis: secuecnias de ADN que no es convertida en otra forma, sino que trabaja in situ, modifivando al ADN al que se encuentra ligao. Regiones del Gen procarionte Tiene una región UT (unidad de transcripción). Y está formado por 4 regiones: Región Codificante (es aquella se codifica). Sitio de iniciación de transcripción. Región Promotora (sitio a partir del cual se regula la expresión de un gen). Región de Termino (donde se termina la transcripción). Se caracteriza por qué no contiene Intrones (regiones no codificantes), solo exones.
- 38. 45 +1 es el sitio donde la polimerasa inserta el primer ribonucleótido para iniciar la transcripción, no justamente donde inicia la traducción a proteína. Región Promotora: está corriente arriba cuando está antes del +1. Regula la transcripción pero no se transcribe. Ct: AUG, UAA, UGA UTR 5: significa que está codificando para mRNA. Sitios del promotor procarionte (región promotora) Se encuentra casi siempre a -65 pares de base corriente arriba.
- 39. 45 La Región Promotora es el sitio en el cual se regula la expresión del gen, mediante inducción o represión que se ejerce sobre la región operadora (O) del gen, mediante Control Negativo. El Control Positivo se ejerce sobre la Caja CRS. Se observan tres tipos de cajas: la Caja CRS, la Caja Prinow (-10) y la Caja CAT (-35),y una región operadora, las cuales son reconocidas por la RNA- Polimerasa bacteriana. Genes bajo control negativo: expresados a menos que sean desactivados por una proteína represora. Una proteína represora se une al ADN para impedir que la ARN pol inicie la transcripción o al ARNm para impedir que se una a un ribosoma para la traducción. Genes bajo control positivo: la expresión solo es posible cuando una proteína reguladora activadora esté presente. Proteína reguladora es esencial para ella, interactia con el ADN y la ARN pol para ayudar a los eventos de la iniciación. Generalmente llamado activador. Estructura de la Región UTR (Del inglés Untranslated Region). Región no traducida por los ribosomas, se transcribe pero no se traduce a proteína. La secuencia S/D (Shine/Dalgarne) es rica en polipurinas y su extremo 5’ en forma de ARNm es complementaria al extremo 3’ del ARNr 16S de la subunidad menor del ribosoma.
- 40. 45 Si requiere péptido de señal o chaperonas, traduce la secuencia líder y produce el péptido de señal. Estructura de un Policistrón (Operón). Se encuentra en los Procariotes en la región comprendida en entre el codón de iniciación (CI) y el codón de terminación (CT) y bajo el control de una misma región promotora. Esta estructura de la región codificante en tandem es llamada Policistrón u Operón debido a que los genes se transcriben juntos bajo el control de una misma región promotora y sus productos se encuentran relacionados metabólicamente, como por ejemplo, el operón lactosa (Lac). Nótese que entre los genes queda una región intergénica corta. Cada cistrón o gen es traducido en tandem por el mismo tipo de ribosoma, previa liberación del polipéptido anterior. Estructura de la región terminal en genes procariotes. Esta se encuentra entre el codón de terminación (CT) y la Señal de terminación de la Transcripción (STT). Estas estructuras son de dos
- 41. 45 tipos: independientes de ρ o dependientes de rho. Las primeras son terminales intrínsecos que no dependen de la subunidad ρ (una proteína hexamérica) para ser reconocidas por la ARN pol y terminar la transcripción. Por el contrario, las STT- ρ dependientes requieren del factor ρ para que la ARN pol puede reconocerlas las asas ρ dependientes, en el contexto del ARNm, y terminar la transcripción. Regiones de los genes eucariontes Existen 3 clases de genes transcritos por el ARN polimerasa I,II,III que codifica en especial para cada gen. Contienen exones que son regiones que codifican una proteína e Intrones que son eliminadas en el proceso ya que son regiones no codificante y que estos ayudan a madurar en ARN. Clase I codificara ARNr Nucleolo ARNP1 Clase II codificara ARNm Nucleoplasma ARNPII Clase III codifica ARNt, ARNnp, ARN5s ARNPIII
- 42. 45 Estructura del gen eucariote – Clase II Estructura del promotor Clase II. Promotor eucariote prototípico mostrando las cajas TATA, CAAT y elementos de respuestas curso arriba. Otros tipos de promotores son hechos exclusivamente de elementos GC o combinaciones de estos con cajas TATA y CAAT.
- 43. 45 Estructura de la región codificante Clase II. Los genes interrumpidos son genes constituidos por una serie alterna de exones e intrones. En eucariotes existen genes con un intron o hasta con más de 100 intrones. En promedio un gen interrumpido humano tiene de 5 a 7 intrones. Los intrones son generalmente más grandes (500pb de longitud) que los exones (100-200pb). El límite exón- intrón esta dado por la presencia de bases consenso GU----…----AG localizadas en los extremos 5’ y 3’ respectivamente de los intrones. Los exones son los que llevan la información codificante para proteína y los intrones no. Una vez es transcrita la región codificante a ARNhn, los intrones son removidos por el “esplaiceosoma” (una serie de ribonucleoproteínas U, RNP-Us: U1, U2, U4, U5 y U6) y los exones empalmados y transportados en forma de ARNm al citoplasma Amplificadores y silenciadores. Son secuencias consenso que intensifican o decrecen la tasa de transcripción de los genes. Estos se localizan generalmente miles de pares de bases curso arriba de un promotor o curso abajo del mismo. En ocasiones pueden estar localizados dentro de un intrón. Estructura del gen eucarionte clase I
- 44. 45 Estructura del gen eucariote Clase I. Los genes clase I generalmente se encuentran en tandem de 1000 a 3000 copias, debido a la necesidad activa de reciclar los ribosomas de la célula eucariota. Estructura del gen eucarionte clase III Estructura del gen eucariote Clase III. Estos genes también se encuentran en tandem en un gran número de copias. La característica más conspícua que los diferencia de los genes clase I y II es que los genes clase III llevan promotores internos dentro de la región codificante (cajas B y C) que da origen al ARNt y cuya función es atraer la ARN pol III, la cual una vez unida a estos, se
- 45. 45 desplaza hacia la caja TATA, esta sí curso arriba de la región codificante, para iniciar la transcripción. Procesamiento del gen Clase III. Una vez transcrito el ARNhn clase I es procesado por ribonucleasas para liberar los ARNt y el ARNr 5S. REGULACIÓN DE GENES 1. Control trascripcional 2. Control del procesamiento del ARN 3. Control del transporte del ARN 4. Control de la traducción 5. Control de la degradación de los ARNm 6. Control de la actividad proteica Operón lac Transcripción es regulada en la fase de inicio, no en la elongación, pero si en la terminación. La traducción también. Dos clases de secuencias de ADN, los que actúan en trans y los que actúan en cis. Genes estructurales bacterianos están organizados en grupos que incluyen genes que codifican para proteínas de función relacionada. Los genes de una vía metabólica están relacionados en grupos. Genes estructurales LacY, Lac Z y Lac A. Lac Z codifica para B-galactosidasa, divide un b-galactosido en sus dos componentes: lactosa a glucosa y galactosa. Lac Y codifica para B-galactósido permeasa, constituye del transporte de b-galactosidos al interior de la celula. Lac A: codifica B-galactosido transacetilasa, transfiere grupo acetilo del acetil CoA a los B- galactosidos. Mutaciones en lac Y y Z: conlleva a Lac que significa pérdida de función. Todo el sistema que incluye los genes estructurales y los elementos que controlan su expresión forman una unidad de regulación que se llama Operón. La act. del operón es regulada por un gen que actúa en forma cis.
- 46. 45 Distinción entre gen estructural y un gen regulador: depende de la mutación. Mutación en estructural: priva a la célula de una proteína particular codificada por el gen. Mutación en reguladores: influye en la expresión de todos los genes estructurales que formen parte de ese grupo. Transcripción de los genes lacZYA es regulada por la expresión del gen Lacl, que aunque está cerca de los estructurales, tiene su propio promotor y propio terminador. Lac l está cerca de la transcripción de los estructurales, pero lacl da de resultado producto transferible por lo q no es necesario esto como tal. Los genes lac son regulados por regulación negativa: transcritos hasta que sean desactivados por proteína reguladora. Mutacion que inactive el regulador: expresión de los lac. Procuto de lacl es el represor lac que se une a región operadora (RP) y a los genes estructurales lacZYA: cuando represor lac una al operador, impide que la ARN pol inicie la transcripción en el sitio promotor. Las bacterias evitan sintetizar las enzimas de una determinada vía, pero aún así están capacitadas para hacerlo en ausencia de nutrientes, rápidamente. INDUCCION: síntesis de enzimas en respuesta a la aparición de un sustrato específico. Transcripción cesa tan rapdo como el inductor sea retirado. Con inductor suficiente las menos de 5 enzimas galactosidasas aumentan a mas de 5000 por cada e.coli. ARN m Lac es inestable y se degrada en menos de 3 minutos: explica la facilidad de ajustarse la bacteria a la retirada de sustrato. REPRESION: si el medio en el que se está desarrollando la bacteria tiene uno de los elementos que sintetiza, (como el W presente en los nutrientes de la e.coli que también sintetiza W) la e. coli detiene su expresión de W. La capacidad de los inductores o represores o moléculas pequeñas no depende de su interacción directa con las enzimas: isopropil tiogalactosido. No es reconocido por la b- galactosidasa, pero es un inductor eficiente los genes lac. Inductores gratuitos: son inductores de enzimas que no son metabolizados, permanecen en su estado natural dentro de la celula. Inductor verdadero sería metabolizado. Ahora….. que componente que responde al inductor es la proteína represora codificada por lacl: el estado del represor determina si este promotor es activado o no::: En ausencia de un inductor, los genes lac ZYA no son transcritos, dado que la proteían represora está en estado activado, unida al operador. Cuando se añade un inductor, el represor es invertido a una forma inactiva que abandona el operador. El represor tiene dos sitios de unión , uno para el operador y otro para la inductora: cambia estructura- CONTROL ALOSTERICO.
- 47. 45 La indicción se lleva a cabo con una regulación coordinada, todos los genes estructurales son expresados al unisono. ARN m se expresa desde 5’: causa la aparición de b.galactosidasa, b-galactosido permeasa y b-galactosido transacetilasa en ese orden. (ZYA) Si el operon está reprimido, siempre puede entrar algo de inductor por otro mecanismo, además existe un nivel basal de expresión del operon. El represor reconoce la sec. De ADN especifaca operador ya que el operador tiene una secuencia palindromica que sirve como medio sitio para el operador. Represion porel catabolito: en e.coli si hay HCO lo va apreferir en lugar de consumir glactosidos. Evita la expresión de operones incluyendo lac, gal y ara. Hay una preferencia por glucosa inhiniendo la expresión de los operones que codifinan las enxiams de vías met. alternativas. la glucosa tiene cap. de reducir cAMP. Manipulación del ADN, ARN y proteínas Extracción del material genético Endonucleasas de restricción Las endonucleasas de restricción son enzimas aisladas de bacterias que cortan el material genético de un virus que puede atacarlas. Sin embargo, existe el problema de que estas enzimas ataquen al mismo material genético de la célula, por lo que este tiene secuencias ocultas en su genoma (metilación en A o C) que impide que las endonucleasas de restricción actúen sobre el ADN propio. Las endonucleasas de restricción reconocen secuencias específicas (4 a 8 nucleótidos). Existen mas de 100 enzimas de este tipo en el mercado, y básicamente su función es purificar genes o ampliar la información. Pueden generar fragmentos cohesivos o fragmentos romos. Estos fragmentos generados podrían unirse con un ADN ajeno y de esa manera formar un ADN recombinate. Si se comparan los tamaños de los fragmentos de restricción generados de una región génica, después del tratamiento con diferentes nucleasas de restricción, se puede construir un mapa de restricción.
