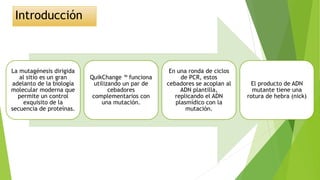
Este documento describe un protocolo modificado de mutagénesis dirigida de un solo paso que permite la eliminación, inserción y mutaciones de sitios múltiples en un plásmido. El método utiliza cebadores de PCR diseñados con secuencias no superpuestas y complementarias entre sí para sintetizar ADN mutante que luego se transforma en células bacterianas. El protocolo modificado se utilizó con éxito para realizar diversas mutaciones, incluidas inserciones y deleciones, en un gen clonado de Staphylococcus