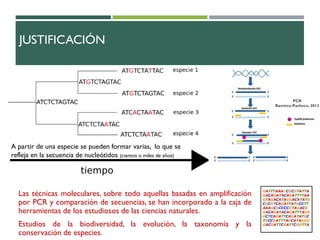
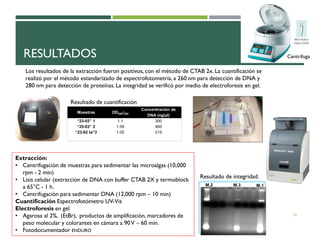
Este documento describe un estudio para identificar microalgas en aguas residuales tratadas mediante la técnica de PCR. Se estandarizó el método de extracción de ADN y amplificación de la región 23S del ADN ribosomal. Se identificaron tres tipos de microalgas mediante el análisis de secuencias nucleotídicas, incluyendo Monoraphidium sp., Mychonastes homosphera y Mychonastes jurissi.




![ANTECEDENTES
5
En ambientes o ecosistemas acuáticos se ha documentado la proliferación de
microalgas a concentraciones de nitrógeno total de 1.2 ppm y de fósforo total de 0.2
ppm [Steven et al., 2012].](https://image.slidesharecdn.com/identificacin-de-microalgas-pcr-230417030822-acd8851a/85/Identificacion-de-microalgas-PCR-pdf-5-320.jpg)