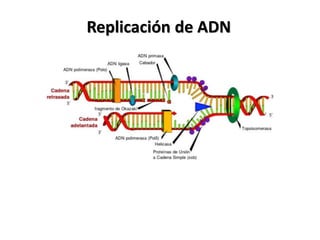
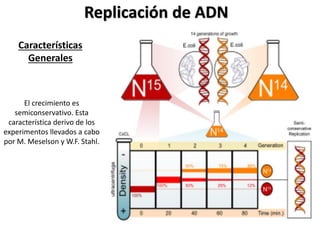
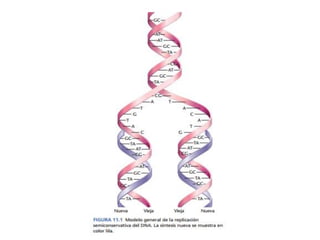
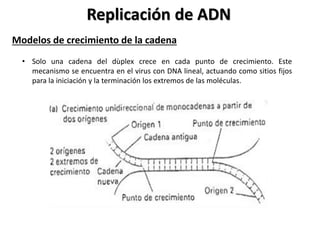
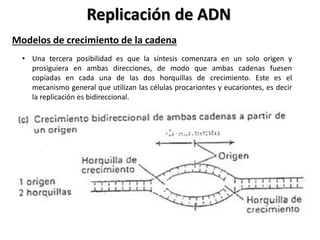
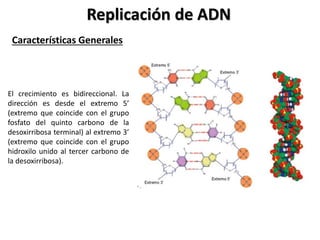
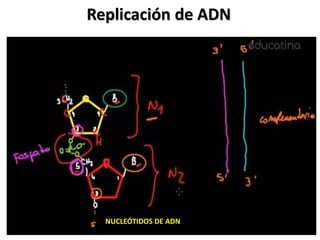
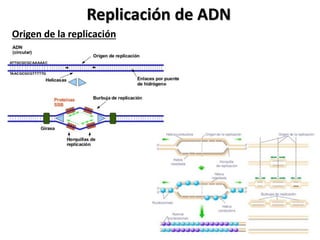
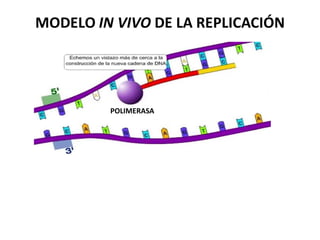
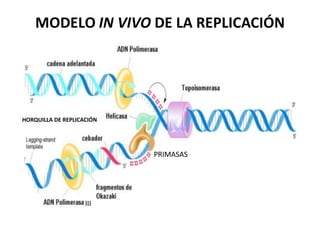
La replicación del ADN es un proceso fundamental en el que se crea una copia idéntica del material genético. Comienza con la iniciación, donde la enzima helicasa desenrolla la doble hélice del ADN, creando una "horquilla" de replicación. Luego, la enzima primasa sintetiza pequeños fragmentos de ARN llamados "primers", que servirán como punto de partida para la síntesis de nuevo ADN. La enzima polimerasa DNA sintetiza nuevo ADN a partir de los primers, utilizando los nucleótidos disponibles en el medio, y también realiza una corrección de errores. Finalmente, la enzima ligasa une los fragmentos de ADN recién sintetizados, formando una cadena continua. Este proceso es esencial para la transmisión de información genética, el crecimiento, desarrollo y reparación de tejidos, y para mantener la estabilidad del genoma.