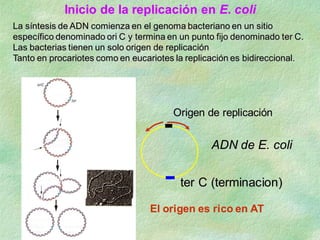
El documento describe el modelo de Watson y Crick de la replicación semiconservativa del ADN, en el que una cadena parental se conserva y sirve como molde para la síntesis de la cadena complementaria. Explica las etapas de iniciación, elongación y finalización de la replicación, incluyendo la apertura de la doble hélice, la síntesis de cebadores de ARN, la polimerización bidireccional y la unión de los fragmentos de Okazaki. También describe la reacción en cadena de la polimerasa (PCR) y sus usos para amplificar segment