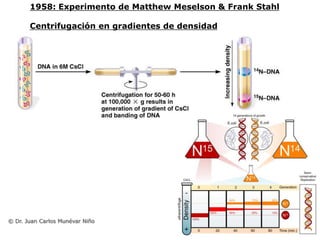
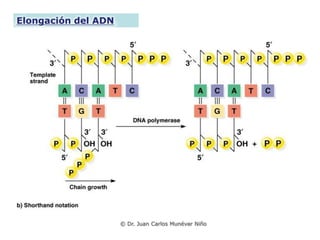
El documento detalla el proceso de replicación del ADN, abordando modelos como el semiconservativo, la función de diversas ADN polimerasas y los mecanismos involucrados en la síntesis y elongación del ADN. Se discuten aspectos clave como la importancia de las proteínas iniciadoras, la helicasa, los cebadores de ARN y el manejo de los telómeros en eucariotas. También se resalta el papel de la actividad exonucleasa en la corrección de errores durante la replicación.