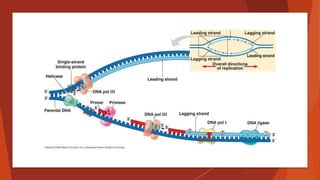
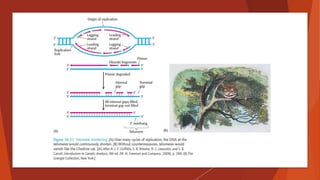
El documento resume los principales aspectos de la replicación del ADN. La replicación es llevada a cabo por enzimas ADN polimerasas que copian fielmente la hebra molde de ADN. Estas enzimas requieren proteínas accesorias y el proceso es semiconservativo, copiando una hebra molde original y una nueva hebra complementaria. La replicación ocurre de forma simultánea en ambas hebras a través de la acción coordinada de enzimas y proteínas asociadas.