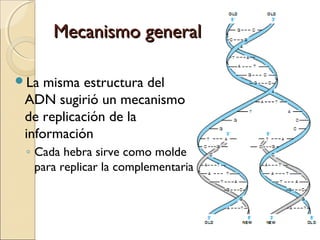
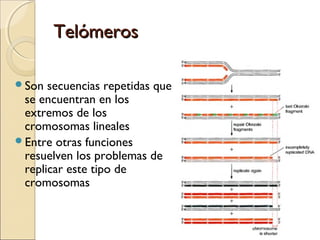
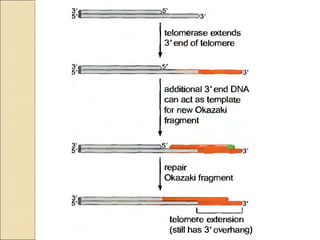
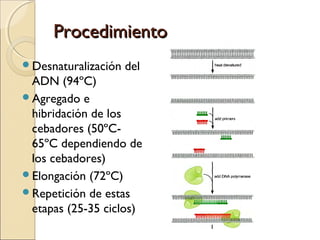
La replicación del ADN es el proceso por el cual el material genético se duplica antes de la división celular. Es semiconservativa, bidireccional y semidiscontinua. En procariotas involucra maquinaria enzimática como ADN polimerasas, helicasas y ligasas. En eucariotas es más complejo con múltiples orígenes de replicación. La replicación in vitro mediante PCR permite amplificar fragmentos de ADN y tiene numerosas aplicaciones.