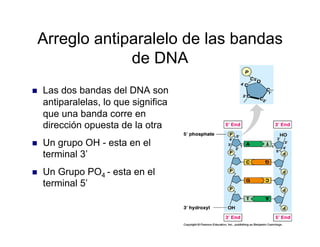
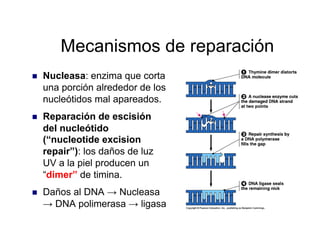
El documento describe los conceptos básicos de la estructura y replicación del ADN. Explica que durante la replicación, la doble hélice del ADN se desenrolla y cada cadena sirve como molde para la síntesis de una nueva cadena complementaria, de modo que cada molécula hija contenga una cadena parental y una nueva. Detalla los mecanismos de la replicación, incluyendo la apertura de la doble hélice por la helicasa, la síntesis de las cadenas continua y discontinua, y la unión de los fragmentos de Okazaki por la ligasa.