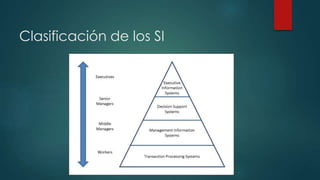
Este documento describe los sistemas de información en bioinformática. Explica que los sistemas de información son conjuntos de elementos como hardware, datos, software y procedimientos que administran información de manera organizada. Luego describe varios tipos de bases de datos bioinformáticas como las de nucleótidos, proteínas y microarrays que almacenan y proveen acceso a enormes cantidades de información genómica y otros datos biológicos. Finalmente, enfatiza que los sistemas de información deben brindar estructuras robustas y flexibles para gest