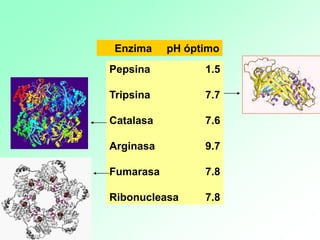
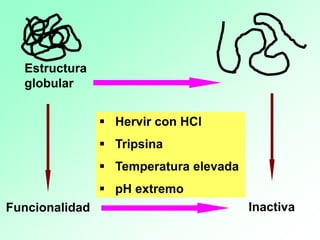
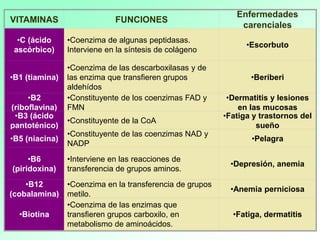
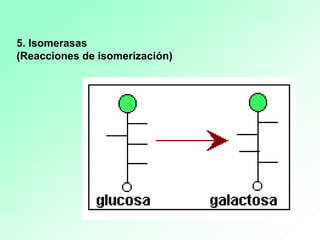
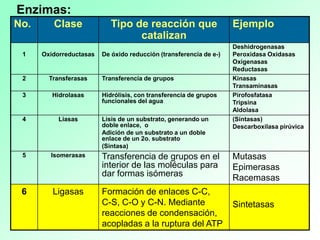
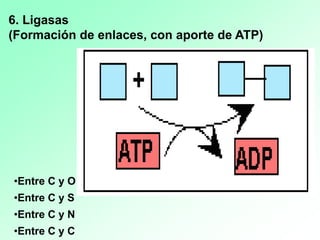
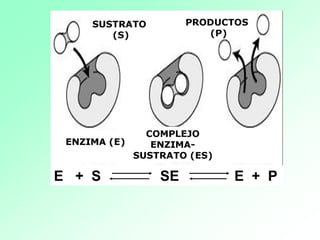
1. Las enzimas son catalizadores biológicos formados por proteínas globulares. 2. Cada enzima es específica para un sustrato particular. 3. Las enzimas aceleran reacciones químicas específicas actuando en disolución acuosa a pH y temperatura óptimos.












![EC 1.1.1.2
Accepted
name:
alcohol dehydrogenase (NADP
+
)
Reaction: an alcohol + NADP
+
= an aldehyde + NADPH + H
+
Other na
me(s):
aldehyde reductase (NADPH2); NADP-alcohol dehydrogenase; NADP
+
-
aldehyde reductase; NADP
+
-dependent aldehyde reductase; NADPH-
aldehyde reductase; NADPH-dependent aldehyde reductase; nonspecific
succinic semialdehyde reductase; ALR 1; low-Km aldehyde reductase;
high-Km aldehyde reductase; alcohol dehydrogenase (NADP
+
)
Systematic name: alcohol:NADP
+
oxidoreductase
Comments: A zinc protein. Some members of this group oxidize only primary
alcohols; others act also on secondary alcohols. May be identical
with EC 1.1.1.19 (L-glucuronate reductase), EC 1.1.1.33
[mevaldate reductase (NADPH)] and EC 1.1.1.55 [lactaldehyde
reductase (NADPH)]. A-specific with respect to NADPH.
Links to other da
tabases:
BRENDA, ERGO, EXPASY, IUBMB, KEGG, PDB, CAS registry
number: 9028-12-0](https://image.slidesharecdn.com/enzimas-240117212939-51037b31/85/Enzimas-caracteristicas-divisiones-pdf-23-320.jpg)











![1. Actuación global de la reacción catalizada.
2. Utilizar un procedimiento analítico para la determinación de S
que desaparece o los productos de la reacción que aparecen.
3. Si la enzima requiere cofactores (iónes metálicos o
coenzimas).
4. La dependencia de la actividad enzimática con la [S] o sea KM
del S.
5. El pH óptimo.
6. Un intervalo de temperatura en la que la enzima es estable y
muestra actividad elevada.
¿Cómo detectar una enzima?](https://image.slidesharecdn.com/enzimas-240117212939-51037b31/85/Enzimas-caracteristicas-divisiones-pdf-41-320.jpg)

