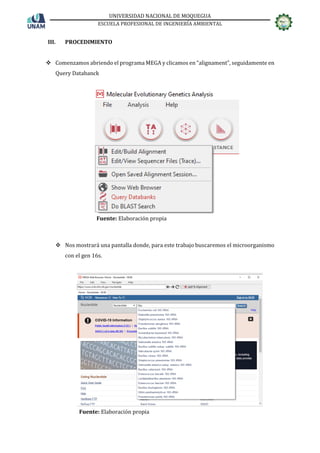
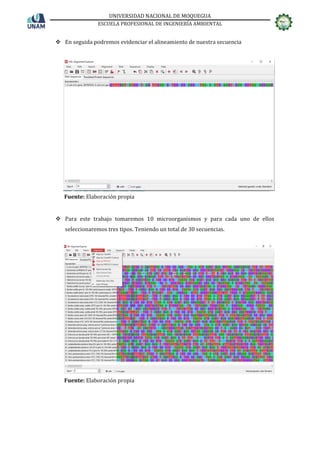
El informe detalla el uso del software MEGA para analizar secuencias de ADN desde una perspectiva evolutiva, destacando sus capacidades avanzadas de alineación de secuencias y construcción de árboles filogenéticos. Además, se describen los pasos para importar microorganismos, alinear secuencias y ejecutar análisis, enfatizando la interfaz modernizada que mantiene familiaridad para los usuarios. El trabajo se basa en el análisis de 30 secuencias de distintos microorganismos incluido en el procedimiento.