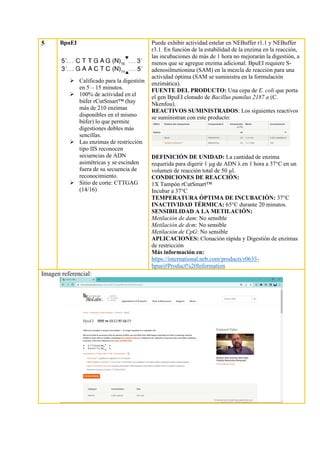
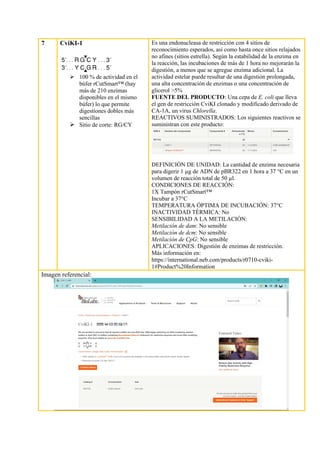
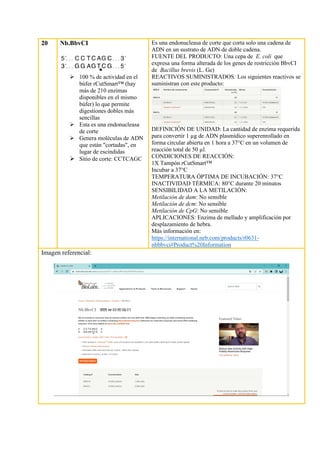
Este documento proporciona información sobre 30 enzimas de restricción. Resume sus características clave como su sitio de corte, fuente, condiciones de reacción y aplicaciones. Explica brevemente los tipos de enzimas de restricción y sus usos en biología molecular, como la clonación y análisis de ADN.