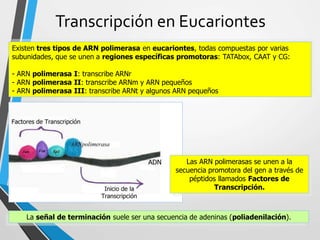
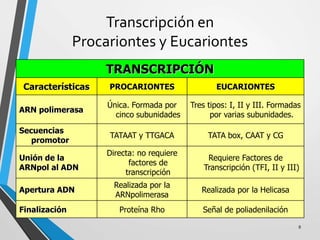
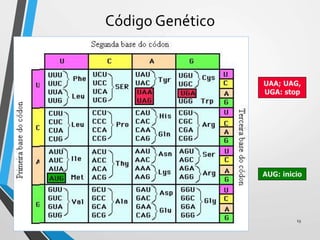
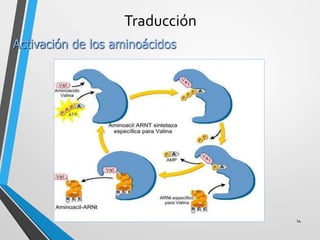
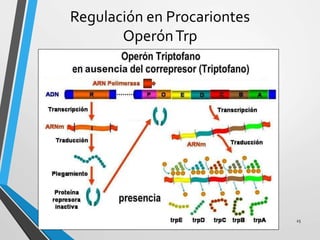
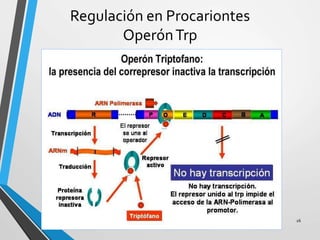
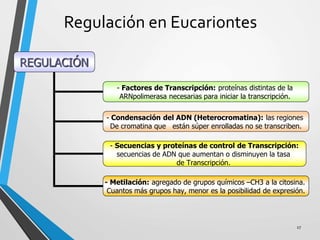
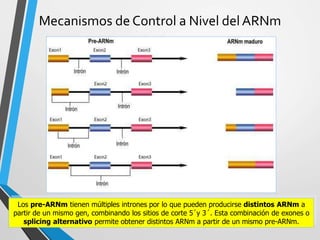
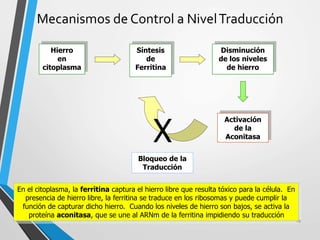
El documento describe los procesos de transcripción, traducción y regulación de la expresión génica. La transcripción convierte la información genética del ADN en ARNm en procariontes y eucariontes. La traducción convierte el ARNm en proteínas. La regulación controla estos procesos a través de operones, factores de transcripción, splicing alternativo, bloqueo de la traducción y marcaje de proteínas para su degradación.