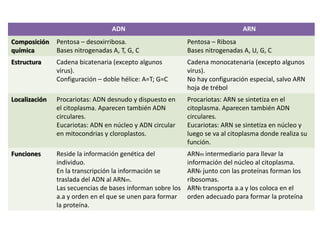
1. El documento describe el proceso de transcripción y traducción, desde la transcripción del ADN al ARN mensajero catalizada por ARN polimerasas, hasta la traducción del ARNm en proteínas por los ribosomas.
2. Se explican las diferencias entre la transcripción en procariotas y eucariotas, como que en eucariotas ocurre en el núcleo y requiere el desenrollamiento de la cromatina.
3. También se describen los diferentes tipos de ARN producidos - ARNm, ARNr y ARNt -