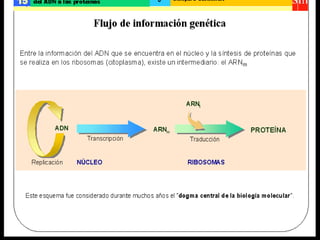
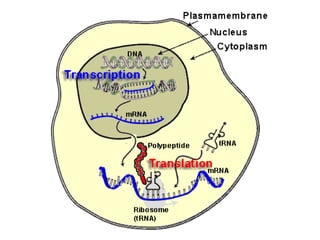
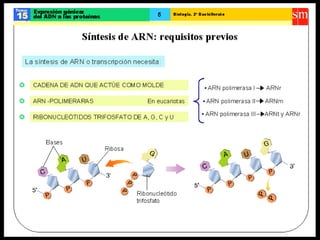
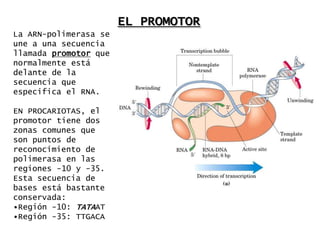
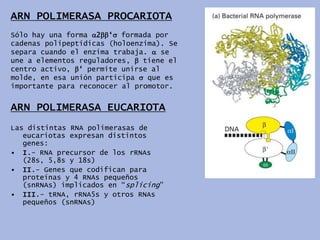
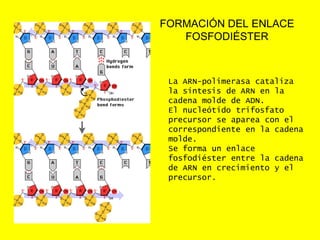
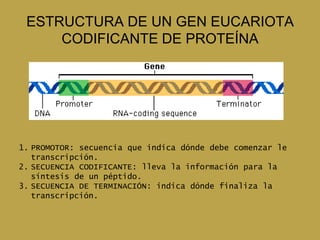
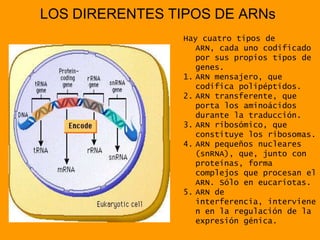
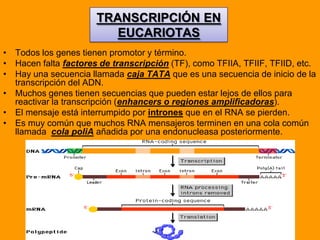
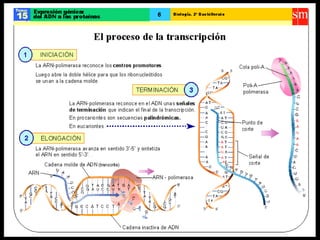
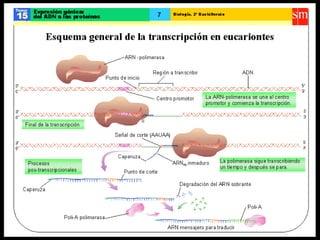
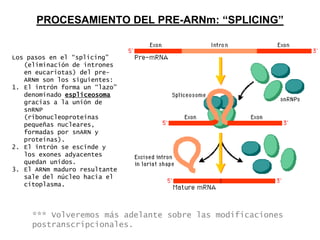
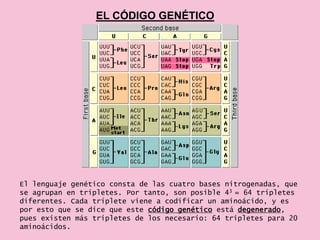
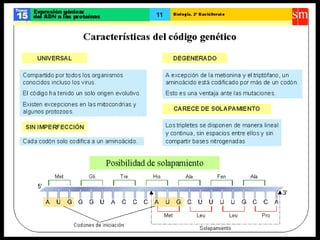
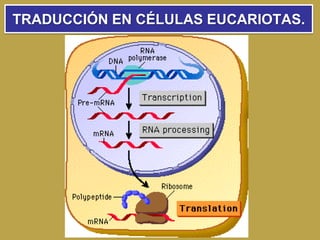
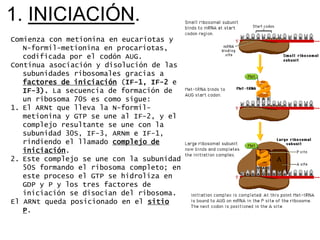
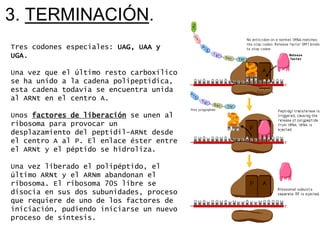
El documento describe el proceso de expresión génica desde la transcripción del ADN al ARNm hasta la traducción del ARNm a proteínas. Explica que durante la transcripción, la ARN polimerasa cataliza la síntesis del ARNm a partir del ADN en el núcleo. Luego, el ARNm sufre modificaciones postranscripcionales como el splicing en eucariotas para quitar intrones. Finalmente, durante la traducción, el ARNm se lee en el citosol por los ribosomas que sintetizan la proteína siguiendo