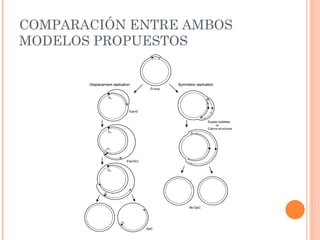
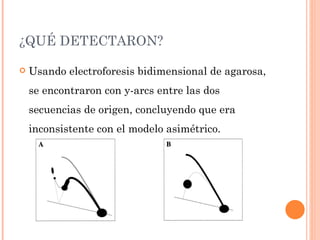
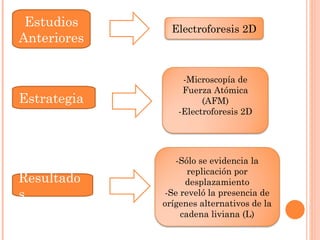
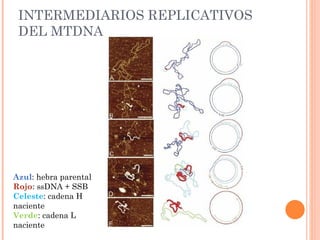
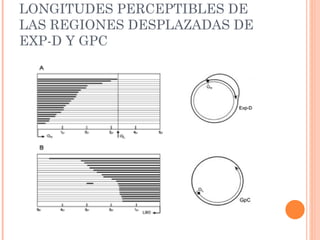
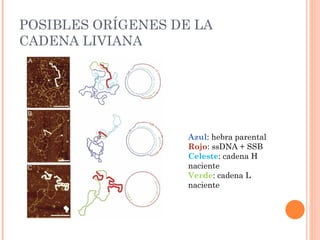
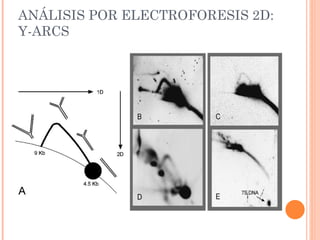
Este documento presenta un estudio que reexamina el modelo de replicación mitocondrial en mamíferos mediante el uso de microscopía de fuerza atómica y electroforesis bidimensional. Los autores encontraron evidencia de orígenes alternativos de la cadena liviana del ADN mitocondrial, lo que contradice modelos previos que proponían una replicación asimétrica. Esto sugiere que la replicación ocurre por desplazamiento de hebras con orígenes alternativos de la cadena liviana, en lugar de un mecanismo acoplado de