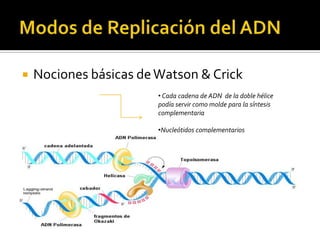
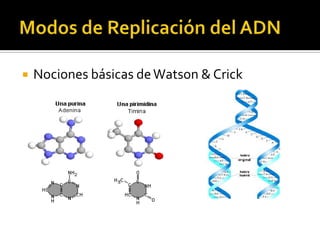
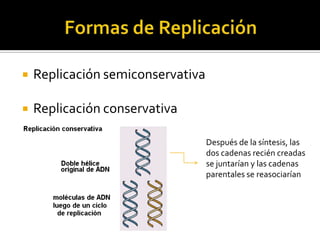
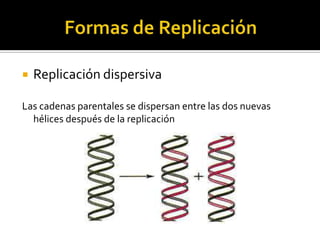
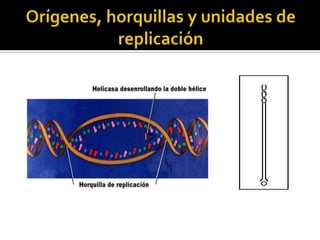
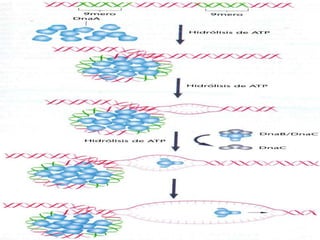
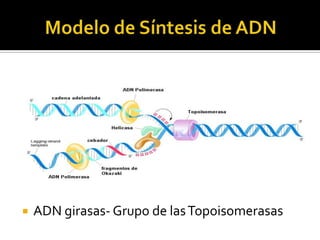
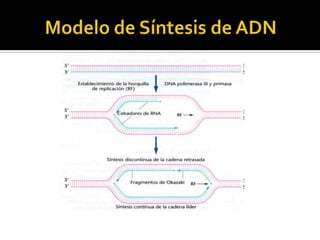
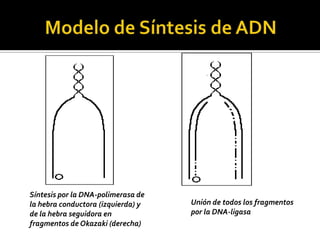
Este documento discute varios conceptos clave relacionados con la replicación del ADN. Explica el modelo de replicación semiconservativa propuesto por Watson y Crick, en el que cada cadena de la doble hélice de ADN sirve como molde para la síntesis de una cadena complementaria nueva. También describe los diferentes tipos de replicación del ADN, incluida la conservativa y la dispersiva, y explica por qué la evidencia apoya el modelo semiconservativo. Además, analiza los mecanismos enzimáticos involucrados en la replicación, como las AD