1.estructura de los acidos nucleicos
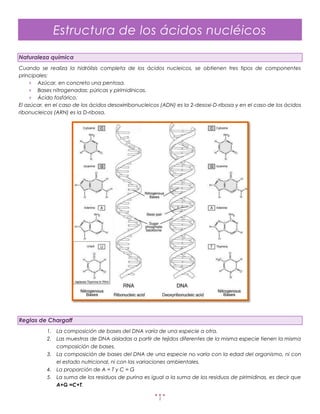
- 1. Estructura de los ácidos nucléicos Naturaleza química Cuando se realiza la hidrólisis completa de los ácidos nucleicos, se obtienen tres tipos de componentes principales: Azúcar, en concreto una pentosa. Bases nitrogenadas: púricas y pirimidínicas. Ácido fosfórico. El azúcar, en el caso de los ácidos desoxirribonucleicos (ADN) es la 2-desoxi-D-ribosa y en el caso de los ácidos ribonucleicos (ARN) es la D-ribosa. Reglas de Chargaff 1. La composición de bases del DNA varía de una especie a otra. 2. Las muestras de DNA aisladas a partir de tejidos diferentes de la misma especie tienen la misma composición de bases. 3. La composición de bases del DNA de una especie no varía con la edad del organismo, ni con el estado nutricional, ni con las variaciones ambientales. 4. La proporción de A = T y C = G 5. La suma de los residuos de purina es igual a la suma de los residuos de pirimidinas, es decir que A+G =C+T. 1
- 2. El modelo de la doble hélice de Watson y Crick El ADN es una doble hélice enrollada helicoidalmente “a derechas” (sentido dextrorso). Enrollamiento de tipo plectonémico: para separar las dos hélices es necesario girarlas como si fuera un sacacorchos. Cada hélice es una serie de nucleótidos unidos por enlaces fosfodiéster en los que un grupo fosfato forma un puente entre grupos OH de dos azúcares sucesivos (posiciones 3’ de un azúcar y 5’ del siguiente). Las dos hélices se mantienen unidas mediante puentes o enlaces de hidrogeno producidos entre las bases nitrogenadas de cada hélice. Siguiendo los datos de Chargaff (1959), la Adenina de una hélice aparea con la Timina de la hélice complementaria mediante dos puentes de hidrógeno. Igualmente, la Guanina de una hélice aparea con la Citosina de la complementaria mediante tres puentes de hidrógeno. Las dos hélices por razones de complementaridad de las bases nitrogenadas son antiparalelas, teniendo secuencias de átomos inversas. Una hélice lleva la secuencia 5’P → 3’ OH , mientras que la hélice complementaria sigue la secuencia de átomos 3’OH → 5’P. Alternativas al modelo de la doble hélice de Watson y Crick El modelo de la Doble Hélice propuesto por Watson y Crick está basado en estudios del ADN en disolución (hidratado). La denominada forma B ó ADN-B tiene un mayor interés biológico ya que es la que presenta el ADN en interacción con las proteínas nucleares. Además de la forma B, existen otras estructuras posibles que puede presentar el ADN. Algunas de estas alternativas son las siguientes: ADN-B: ADN en disolución, 92% de humedad relativa, se encuentra en soluciones con baja fuerza iónica se corresponde con el modelo de la Doble Hélice. 10 pb por giro ADN-A: ADN con 75% de humedad, requiere Na, K o Cs como contraiones, presenta 11 pares de bases por giro completo y 23 Å de diámetro. Es interesante por presentar una estructura parecida a la de los híbridos ADN-ARN y a las regiones de autoapareamiento ARN-ARN. ADN-C: ADN con 66% de humedad, se obtiene en presencia de iones Li, muestra 9+1/3 pares de bases por giro completo y 19 Å de diámetro. 2
- 3. ADN-Z: doble hélice sinistrorsa (enrollamiento a izquierdas), 12 pares de bases por giro completo, 18 Å de diámetro, se observa en segmentos de ADN con secuencia alternante de bases púricas y pirimidínicas (GCGCGC), debido a la conformación alternante de los residuos azúcar-fosfato sigue un curso en zig-zag. Requiere una concentración de cationes superior a la del ADN-B, y teniendo en cuenta que las proteínas que interaccionan con el ADN tienen gran cantidad de residuos básicos sería posible que algunas convirtieran segmentos de ADN-B en ADN-Z. Las posiciones N7 y C8 de la Guanina son más accesibles. ADN con enrollamiento paranémico: Las dos hélices se pueden separar por traslación, cada hélice tiene segmentos alternantes dextrorsos y sinistrorsos de unas cinco bases. Uno de los principales problemas del modelo de la doble hélice (ADN-B) es el enrollamiento plectonémico, para separar las dos hélices es necesario girarlas como un sacacorchos, siendo necesario un gran aporte energético. ADN triple hélice o ADN-H: "In vitro" es posible obtener tramos de triple hélice intercalando oligonucleótidos cortos constituidos solamente por pirimidinas (timinas y citosinas) en el surco mayor de una doble hélice. Este oligonucleótido se une a pares de bases A-T y G-C mediante enlaces de hidrógeno tipo Hoogsteen que se establecen entre la T o la C del oligonucleótido y los pares A-T y G-C de la doble hélice. No se sabe la función biológica del ADN-H aunque se ha detectado en cromosomas eucarióticos. ADN cuadruplexo: "In vitro" se han obtenido cuartetos de Guanina (ADN cuadruplexo) unidas mediante enlaces tipo Hoogsteen, empleando polinucleótidos que solamente contienen Guanina (G). Los extremos de los cromosomas eucarióticos (telómeros) tienen una estructura especial con un extremo 3' OH de cadena sencilla (monocatenario) en el que se repite muchas veces en tandem una secuencia rica en Guaninas. Se piensa que el ADN cuadruplexo telomérico serviría para proteger los extremos cromosómicos de la degradación enzimática. Ejemplo de secuencia telomérica rica en guaninas (G): 5´P TTGGGTTGGGGTTGGGG...............TTGGGG 3'OH Fuerzas presentes en la doble hélice Enlace de H con poca contribución (fuerza entrópica) Apilamiento de bases (fuerza entálpica) Interacciones hidrofóbicas (fuerza entálpica) Interacciones iónicas de repulsión de fosfatos y la presencia de cationes que actúan como contra iones estabilizando el DNA (los cationes divalentes son más eficientes que los monovalentes – el Mg2+ estabiliza la estructura del RNA). 3
- 4. Densidad de los ácidos nucleicos Densidad: existe una relación lineal entre el contenido en G+C y la densidad del ADN determinada en un gradiente de densidad. A mayor contenido en G+C mayor densidad posee el DNA esto se debe al apilamiento e interacción hidrofóbica de las bases. Cuando se centrifuga a alta velocidad un solución densa (saturada) de un soluto de bajo peso molecular (ClCs 7,7 M, sucrosa, etc.) se produce un equilibrio entre dos fuerzas opuestas, la de difusión del soluto y la fuerza de sedimentación, como consecuencia se produce un gradiente de densidad que aumenta en la dirección de la fuerza centrifuga (aumenta a medida que avanzamos hacia el fondo del tubo de centrifuga). Si añadimos al centrifugar una molécula de ADN, está migrará hacia el fondo del tubo hasta llegar al punto en que la densidad del soluto de bajo peso molecular (la densidad del ClCs) coincide con la densidad del ADN centrifugado (densidad de flotación). Desnaturalización del DNA Desnaturalización: la proporción A+T/C+G está relacionada en primer lugar con la estabilidad de la molécula de ADN de doble hélice. Cuanto mayor es el contenido en G+C de una molécula, mayor cantidad de pares G-C presentará, como consecuencia tendrá una mayor cantidad de triples enlaces y, por consiguiente, será necesario suministrar una mayor cantidad de energía a esa doble hélice para separar sus dos hebras (desnaturalización o fusión del ADN). Cuanto mayor es el contenido en G+C mayor cantidad de calor que hay que suministrar a un ADN de doble hélice para desnaturalizarlo. La temperatura de fusión o melting (Tm) necesaria para desnaturalizar la mitad del ADN de una mezcla está directamente relacionada con el contenido en G+C, a mayor contenido en G+C mayor temperatura de fusión (Tm). 4
- 5. ¿Cómo se mide? La desnaturalización puede verificarse midiendo la absorbancia de la luz UV que atraviesa una solución con DNA. El DNA absorbe a 260 nm; cuando se encuentra como dsDNA, absorbe un 40% menos que cuando se encuentra como ssDNA porque el efecto de apilamiento entre las bases disminuye la capacidad de absorción. (ssDNA efecto hipercrómico) Efecto de la fuerza iónica Al aumentar la fuerza ionica (concentración de Na) aumenta la Tm porque esta aisla las cargas negativas de los fosfatos estabilizando la doble hélice Efecto de la concentración del DNA Si variamos la concentración del DNA no tiene ningún efecto sobre la Tm porque siempre hay el mismo porcentaje de GC, solo varia en el volumen en el que estaría disuelto ¿Cómo se pone en manifiesto? Utilizando agentes desnaturalizantes como: Variar la fuerza iónica afectando a las bases y favoreciendo la repulsión de los fosfatos. Destruir la estructura del agua utilizando agentes caotrópicos y por lo tanto afectando las fuerzas hidrofóbicas. Aumentando el pH para desproteger a las bases e incentivar la tautomerización. Introducir agentes formadores de puentes de hidrogeno para desfavorecer la unión entre bases. Aumentar la temperatura para romper todas las fuerzas entálpicas. Absorbancia del DNA Absorbancia a 260nm: El estado físico de los ácidos nucleicos está relacionado con su capacidad de absorción de la luz ultravioleta (UV) a 2.600 Å. El menor grado de absorción se produce en estado de doble hélice, la absorción aumenta cuando se produce la desnaturalización pasando a estado de hélice sencilla (efecto hipercrómico, aumento de la absorbancia) y, por último, si degradamos este ADN de hélice sencilla a nivel de nucleótidos libres, de nuevo aumenta la absorbancia. Nucleótido Doble s libres Doble hélice hélice sencilla 5 EFECTO HIPERCRÓMICO
- 6. Renaturalización del DNA La renaturalización es un proceso que depende del estado de desnaturalización. Cuando las hebras no están completamente separadas, es un proceso muy rápido, pero deben existir al menos 12 residuos apareados por complementariedad. Si las dos hebras ya estaban completamente separadas, el proceso se da en dos pasos. En el primero, y relativamente lento, se encuentran las cadenas y se unen por complementariedad en una pequeña secuencia. El segundo paso, más rápido, las bases se aparean sucesivamente como una “cremallera” para formar la doble hélice. La temperatura de renaturalización se define como: Trenaturalización= Tm – 20°C La etapa limitante es la nucleación ya que depende de la frecuencia de choque de las moléculas y esta depende de la concentración de las mismas. Cinética de renaturalización, curvas Cot Velocidad de renaturalización: la velocidad de renaturalización del ADN de un organismo está relacionada con su complejidad. La complejidad se define como la suma del número de nucleotidos que tiene cada tipo de secuencia sin tener en cuenta el número de veces que esta repetida. El ADN de los virus y las bacterias en su mayoría sólo tiene secuencias que están una sola vez en el genoma (secuencias únicas). Sin embargo, los organismos más complejos, como los eucariontes, presentan distintos tipos de secuencias en su genoma. Los eucariontes tienen secuencias únicas (SU), secuencias de bajo número de copias (SBNC, 2-10 copias), secuencias repetidas (SR, alrededor de 102 copias) y altamente repetidas (SAR, 103 a 106 copias). Distintos tipos de secuencias que se observan en el ADN de organismos eucarióticos Genes o secuencias de copia única. En general, la mayor parte de los genes que codifican para polipéptidos, los llamados genes estructurales, se encuentran en una sola copia en el genoma. Secuencias moderadamente repetidas. Se trata de genes o secuencias funcionales que codifican para proteínas relacionadas encuentran generalmente dispersas en el genoma, intercaladas en los brazos cromosómicos, lo que da origen a una nueva clasificación, en secuencias intercaladas cortas (SINES) o largas (LINES). Secuencias altamente repetidas. Las secuencias altamente repetidas se encuentran agrupadas en sectores cromosómicos definidos, generalmente alrededor del centrómero o de los telómeros de los cromosomas. 6
- 7. Cot ½ es el valor de Cot cuando se reasocia el 50% del DNA A mayor complejidad (tamaño), aumenta Cot ½ La re asociación depende de la probabilidad de que una secuencia encuentre a su complementaria por lo tanto: A mayor complejidad (tamaño) aumenta Cot ½ y aumenta la velocidad de renaturalización Al aumentar el número de secuencias repetidas, aumenta la probabilidad de re asociación y aumenta Cot ½ (Ej. DNA satélite) La velocidad disminuye al aumentar el % de bases mismatch y por lo tanto disminuye Cot ½ Las curvas de renaturalización o curvas Cot, de organismos que carecen de secuencias repetidas como virus y bacterias, tienen forma de S, tienen un sólo punto de inflexión o punto medio de la reacción. Todas las secuencias son únicas y tienen la misma probabilidad de encontrar a su complementaria para formar la doble hélice. En cambio en eucariotas, la complejidad es mucho mayor porque existen diferentes secuencias y cada una de ellas esta repetida. Por lo tanto la velocidad de renaturalización del DNA aumenta con respecto a procariotas. Para poder realizar el experimento y medir las concentraciones en el tiempo, se utiliza la propiedad de absorbancia a 260 nm. La A260 de un DNA responde a: Se puede medir la diferencia de los picos ss y de ds para ver la relación C/Co. Para separar el dsDNA del ssDNA, se puede realizar una cromatografía en columna usando hidroxiapatita, un compuesto que es más afín por el DNA de simple cadena. De manera que eluye primero el DNA de doble cadena cuando se lava la columna con una solución de hidróxido de sodio. A partir de la segunda ecuación planteada se puede observar que: 7
- 8. Es decir que para 2 DNA distintos estamos renaturalizando en las mismas condiciones, nos permite hallar su longitud. K es proporcional a la complejidad del DNA y está relacionada con el factor N (que es la “longitud” de la molécula de DNA). Se define a N como el número de nucleótidos de secuencias no repetidas, de donde: De donde: Dividiendo: Para una secuencia poli dA-dT, el número de complejidad es 1, dado que no hay secuencias repetidas, para una secuencia del tipo ACGTTAG el número de complejidad es 7, porque hay 7 diferentes nucleótidos. Parámetro Efecto sobre Tm Efecto sobre la velocidad de renaturalización Composición de Incremento de Tm con el aumento del %G-C no ejerce efecto bases Longitud <150 bp; incremento de Tm con el incremento de Incrementa la velocidad con la longitud; >500 bp no hay efecto longitud Fuerza ióncia Incremento de Tm con el aumento de [Na+] optimo a 1.5M Na+ %bp mismatch Disminuye Tm con el aumento de %mismatch Disminuye la velocidad con el aumento de %mismatch Concentración no ejerce efecto Aumenta la velocidad con el aumento de [DNA] Agente disminuye Tm wcon el aumento de [formamide], optimo a 50% formamide denaturalizante [urea] Temperatura - optima at 20°C por debajo de Tm Estructura del Cromosoma Eucariota En el caso de los organismos eucariontes el cromosoma nace fundamentalmente de la interacción entre el ADN, las histonas y las proteínas no histónicas. Los cromosomas eucarióticos son moléculas muy largas de ADN doble hélice en interacción con proteínas (histonas y no histonas) que se pueden encontrar desde estados relajados o poco compactados como en los núcleos de las células en interfase hasta en estados altamente compactados como sucede en la metafase mitótica. 8
- 9. Estructura del Cromosoma del los Virus El material hereditario de los virus está organizado en cromosomas de diferente tipo. Desde el punto de vista genético, los virus pueden clasificarse en virus ADN o ARN, doble hélice o hélice sencilla, y en circular o lineal. Virus cuyo material hereditario es ADN. Virus cuyo material hereditario es ARN. Virus cuyo material hereditario es ARN-ADN. Virus cuyo material hereditario es ADN-ARN. ¿Cómo armamos un cromosoma artificial de levadura? ARS1: secuencia de replicación autónoma CEN4: centrómero del cromosoma IV, permite la segregación del plásmido durante la división celular TELs: secuencias teloméricas. HIS3, TRP1, URA3, TRP1: marcadores de selección en levadura confieren prototrofía para histidina, triptófano y uracilo 9
