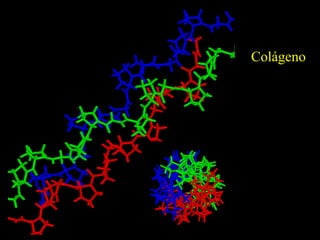
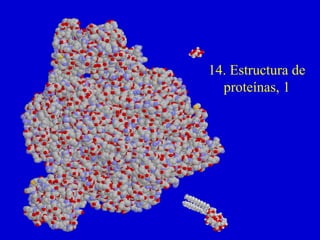
El documento describe los diferentes niveles estructurales de las proteínas, incluyendo la estructura primaria (secuencia de aminoácidos), secundaria (plegamiento básico debido a enlaces de hidrógeno), terciaria (estructura tridimensional) y cuaternaria (asociación de subunidades). También presenta ejemplos de técnicas para determinar la estructura primaria como la secuenciación del ADN y la digestión enzimática.


















![Predicciones a partir de estructura primaria, 2: Homologías
( C G 5 4 3 2 ) C G 5 4 3 2 P R O T E I N . [ D r o s o p h i l a m e l a n o g a s t e r ]
3 7 6 A A
S c o r e = 3 9 8 b i t s ( 1 0 1 1 ) , E x p e c t = e - 1 1 0
I d e n t i t i e s = 1 9 7 / 3 5 5 ( 5 5 % ) , P o s i t i v e s = 2 4 5 / 3 5 5 ( 6 8 % ) , G a p s = 2 / 3 5 5 ( 0 % )
Q u e r y : 2 Y Q Y P A L T P E Q K K E L S D I A H R I V A P G K G I L A A D E S T G S I A K R L Q S I G T E N T E E N R R F Y R Q L 6 1
+ Y P E + + E L I + + V A P G K G I L A A D E S + + K R Q I G E N T E E N R R Y R Q +
S b j c t : 5 F Y Y P - - N K E L Q E E L I C I S K A L V A P G K G I L A A D E S S A V M G K R F Q L I G V E N T E E N R R L Y R Q M 6 2
Q u e r y : 6 2 L L T A D D R V N P C I G G V I L F H E T L Y Q K A D D G R P F P Q X X X X X X X X X X X X X X X X X X P L A G T N G E 1 2 1
L T D + + I G V I + H E T L + Q + D D G P F + P L G + E
S b j c t : 6 3 L F T T D P K I A E N I S G V I F Y H E T L H Q R T D D G L P F V E A L R K K G I L T G I K V D K H F S P L F G S E D E 1 2 2
Q u e r y : 1 2 2 T T T Q G L D G L S E R C A Q Y K K D G A D F A K W R C V L K I G E H T P S A L A I M E N A N V L A R Y A S I C Q Q N G 1 8 1
T T Q G L D L + R C A Q Y K K + G F A K W R C + L K I + + T P S A I + E N A N V + A R Y A + I C Q
S b j c t : 1 2 3 F T T Q G L D D L A N R C A Q Y K K E G C S F A K W R C I L K I T K N T P S P Q A I L E N A N V M A R Y A A I C Q S Q R 1 8 2
Q u e r y : 1 8 2 I V P I V E P E I L P D G D H D L K R C Q Y V T E K V L A A V Y K A L S D H H I Y L E G T L L K P N M V T P G H A C T Q 2 4 1
+ V P I + P E + L G D H D L R C Q V E + L A V Y K A L S D H H + + L E G T L L + P + M V P G +
S b j c t : 1 8 3 L V P I I S P E V L A T G D H D L D R C Q K V N E I L L A G V Y K A L S D H H V F L E G T L L Q P S M V M P G L Q S N K 2 4 2
Q u e r y : 2 4 2 K F S H E E I A M A T V T A L R R T V P P A V T G I T F L S G G Q S E E E A S I N L N A I N K C P L L K P W A L T F S Y 3 0 1
+ I + A T V A + R R + V P P A V G + F G Q S E E E A + + + L N A I N P L K P W A + T F + +
S b j c t : 2 4 3 N H P P A D I G V A T V L A I R R S V P P A V M G V L F C G G A Q S E E E A T V H L N A I N N V P L C K P W A M T F A F 3 0 2
Q u e r y : 3 0 2 G R A L Q A S A L K A W G G K K E N L K A A Q E E Y V K R A L A N S L A C Q G K Y T P S G Q A G A A A S E S L 3 5 6
R A L Q S L + W G G K K E + A Q E + K R A N L A G K Y + A A + E L
S b j c t : 3 0 3 D R A L Q T S I L R T W G G K K E Q I S H A Q N E L I K R C R A N G L A S I G K Y V I G S V E S S A A T E R L 3 5 7](https://image.slidesharecdn.com/estructuraproteinas-141126102450-conversion-gate02/85/Estructura-proteinas-20-320.jpg)

![Algunos motivos secuenciales en las proteínas
N-Glicosilación N-{P}-[ST]-{P}
Unión a glicosaminoglicano S-G-x-G
Fosforilación dependiente de cAMP [RK]-(2)-[ST]
Fosforilación, protein kinasa C [ST]-x(2)-[RK]
Fosforilación, tirosin kinasa [RK]-x(2)-[DE]-x(3)-Y
N-miristilación G- {EDRKHPFYW}-x(2)-[STAGCN]-{P}
Amidación C-terminal x-G- [RK]-[RK]
g-carboxilación de ácido glutámico x(12)-E-x(3)-E-x-C-x(6)-[DEN]-x-[LIVMFY]
Prenilación C-{DENQ}-[LIVM]](https://image.slidesharecdn.com/estructuraproteinas-141126102450-conversion-gate02/85/Estructura-proteinas-22-320.jpg)