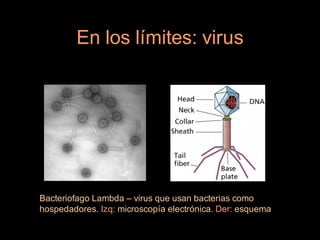
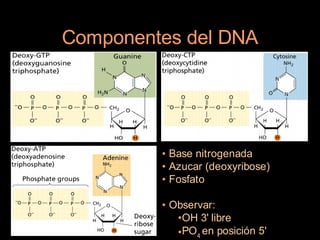
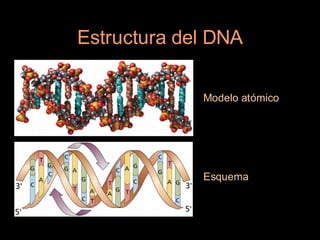
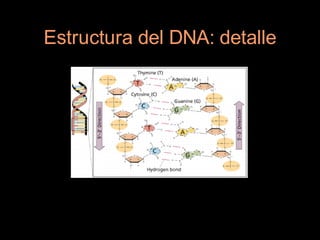
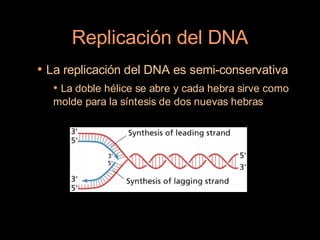
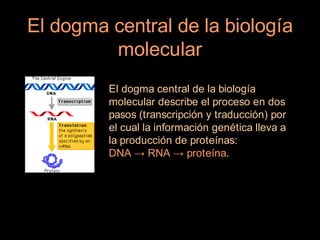
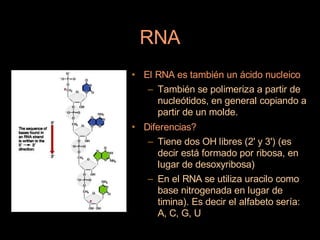
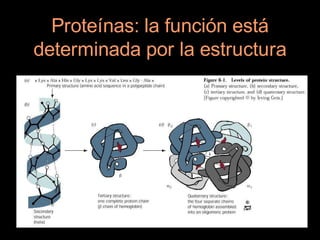
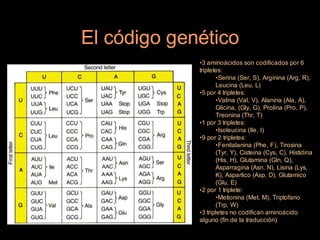
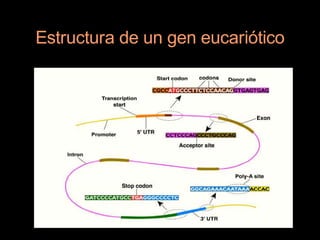
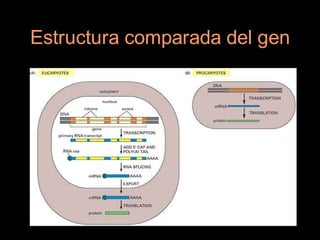
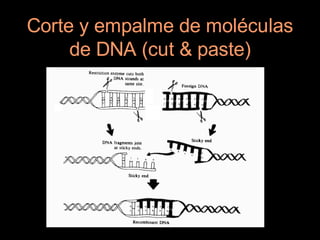
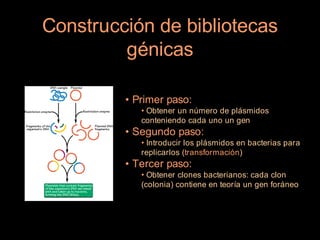
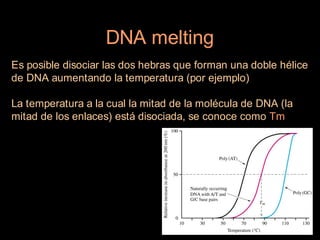
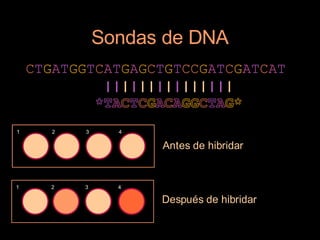
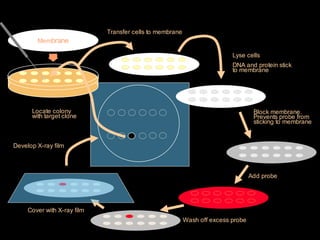
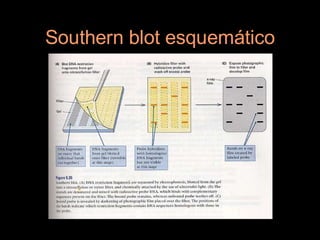
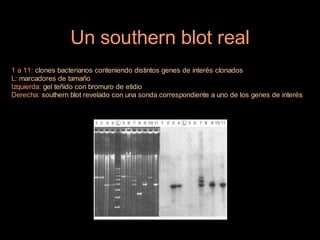
Este documento proporciona una introducción a conceptos básicos de biología molecular para científicos de computación interesados en bioinformática. Explica brevemente las características centrales de los seres vivos, la herencia genética a través del ADN y ARN, y cómo la información genética se transmite en dos pasos para producir proteínas a través de la transcripción y traducción. También describe conceptos clave como la estructura del ADN y ARN, la replicación, transcripción y traducción, y técnic