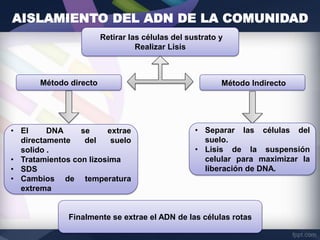
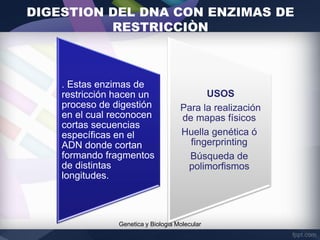
La técnica de T-RFLP involucra 5 pasos principales: 1) aislamiento de ADN de la comunidad microbiana, 2) amplificación del gen 16S rRNA a través de PCR, 3) digestión del ADN amplificado con enzimas de restricción, 4) separación electroforética de los fragmentos de restricción terminales, y 5) análisis comparativo de los perfiles de fragmentos para estudiar la estructura y dinámica de la comunidad microbiana.