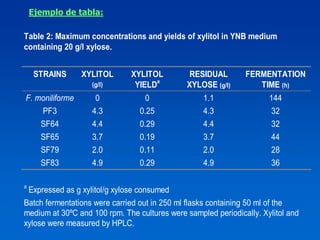
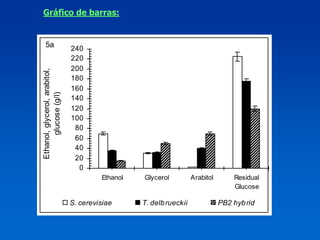
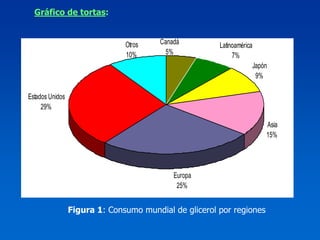
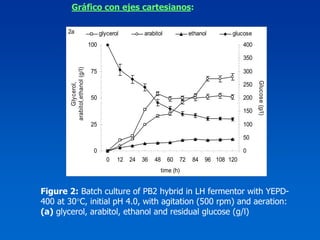
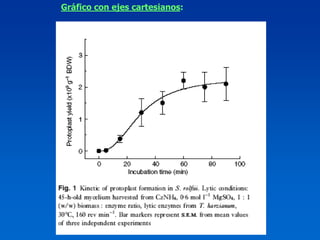
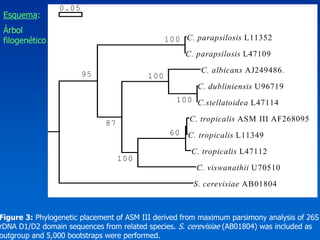
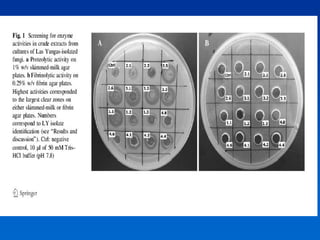
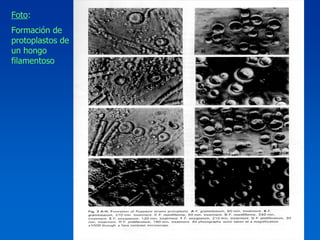
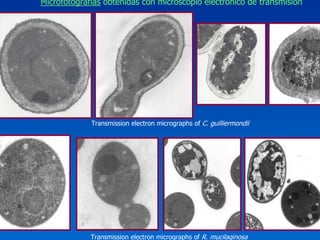
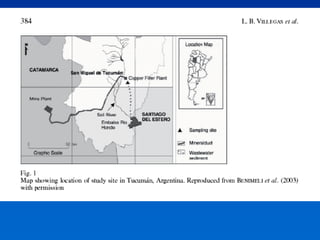
El documento presenta un taller sobre la metodología de la investigación científica. Explica que la estructura de un trabajo científico experimental incluye el título, autores, introducción, antecedentes, materiales y métodos, resultados, discusión, conclusiones y bibliografía. Además, detalla los pasos para la comunicación del trabajo científico, incluyendo la estructura, contenido y estilo de los resúmenes y manuscritos, así como las normas para presentaciones en congresos y publicaciones.