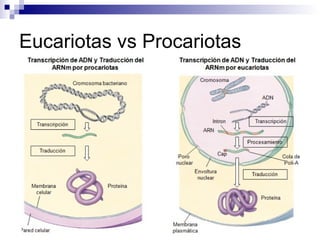
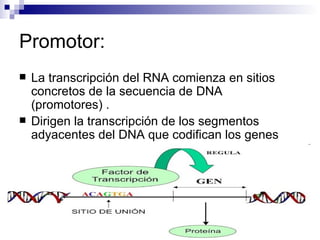
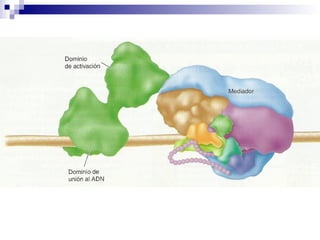
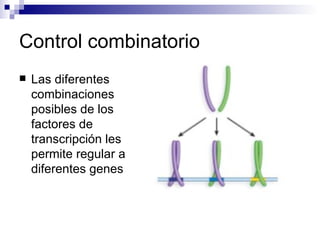
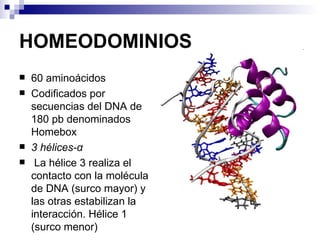
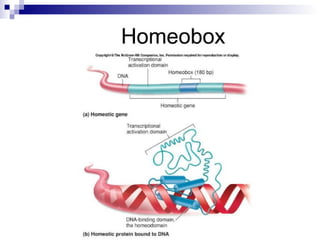
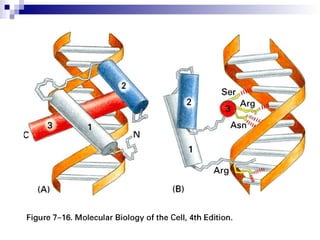
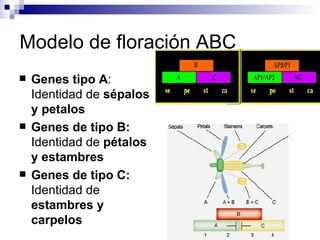
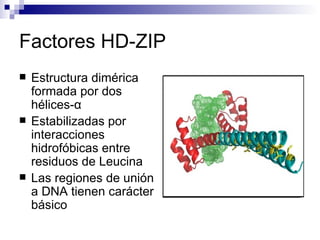
El documento aborda los factores de transcripción en plantas, sus funciones y estructuras, así como su papel en el desarrollo fenotípico en respuesta a señales ambientales. Se centra en modelos como Arabidopsis thaliana y describe diferentes tipos de factores de transcripción, incluyendo homeodominios y mad box, y su función en la regulación de genes. También se discuten mutaciones en genes específicos que afectan el desarrollo de órganos florales y la interacción entre desarrollo y señales ambientales.