Biología Molecular
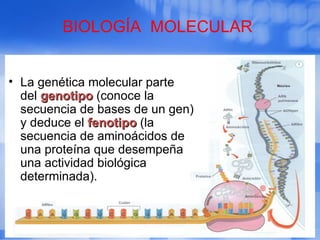
- 1. • La genética molecular parte del genotipogenotipo (conoce la secuencia de bases de un gen) y deduce el fenotipofenotipo (la secuencia de aminoácidos de una proteína que desempeña una actividad biológica determinada). BIOLOGÍA MOLECULAR
- 2. Dogma central de la biología molecularDogma central de la biología molecular • El ADN se replicareplica y heredamos copias del ADN paterno y materno (el genotipo), en cuyas moléculas se localiza los genes. Estos poseen mensajes codificados y, cuando se expresan (se transcribentranscriben y traducetraduce) se forman las diversas proteínas que ponen de manifiesto los caracteres heredados (el fenotipo)
- 4. Contexto histContexto históricoórico • La primera prueba de que los genes estaban formados por ADN proceden de los experimentos de transformación bacteriana realizados por el bacteriólogo F Griffith en 1928. • Con este este experimentoexperimento Griffith demostró que existía un factor transformantefactor transformante
- 5. • Las cepas de bacterias virulentas (Streptococcus pneumoniae) que provocan la muerte del ratón por neumonía se designan con la letra SS.
- 6. • Las cepas inocuas no provocan la muerte del ratón y se designan con la letra RR.
- 7. • Las bacterias S, calentadas y destruidas por el calor no provocan neumonía.
- 8. • La inyección de una mezcla de bacterias S destruidas por el calor y bacterias y R produce la muerte del ratón. • Los cultivos de bacterias S destruidas por el calor contienen el factorfactor transformantetransformante (“ADN”) que transforma a algunas bacterias inocuas R en bacterias virulentas S
- 9. • En 1944, los investigadores Avery, Mcleod y McCarthy identificaron que el factor transformante descubierto por Griffith era el ADNADN. • Sin embargo una buena parte de la comunidad científica permaneció escéptica y hubo que esperar hasta 1952, cuando Alfred Hershey y Martha Chase confirmaron experimentalmente y de manera irrefutable que es el ADN y no las proteínas la molécula portadora de la información genética.
- 10. La transcripción en eucariotasLa transcripción en eucariotas • Es la primera fase de la expresión génica, durante la cual se transfiere la información del ADN al lenguaje del ARN. Este proceso consiste en la síntesis de una molécula de ARN. • En eucariotas, todos los tipos de ARN (ARNm, ARNr y ARNt) se sintetizan en el núcleo y luego se exportan al citoplasma, a través de los poros nucleares.
- 11. Elementos que intervienen en laElementos que intervienen en la ssííntesis del ARNmntesis del ARNm • Una cadena de ADN que actUna cadena de ADN que actúúa como molde.a como molde. • RibonucleRibonucleótidos.ótidos. • Una enzima: el ARN polimerasa II o transcriptasa.Una enzima: el ARN polimerasa II o transcriptasa.
- 12. Cadena de ADN que actCadena de ADN que actúúa como moldea como molde • Hace falta un modelo o patrón y para ello se utiliza una de las dos cadenas del gen que se va a transcribir. • La cadena que se utiliza como modelo se denomina hebra molde y a la otra hebra informativa. • La hebra molde siempre se lee en sentido 3´- 5´y la cadena de ARN transcrito va creciendo en sentido 5´- 3´.
- 13. RibonucleRibonucleótidosótidos • Hacen falta ribonucleótidos de A, G, C y U que estén activados, es decir, en sus formas trifosfato “cargados” de energía: ATP, GTP, CTP y UTP. • La síntesis de ARNm no es más que un proceso de unión de nucleótidos mediante enlaces éster, que se establecen entre el ácido fosfórico situado en posición 5´ de cada nucleótido y el grupo -OH en posición 3´ del último nucleótido de la cadena.
- 14. ARN polimerasa II o transcriptasaARN polimerasa II o transcriptasa • Cataliza la polimerización de los ribonucleótidos basándose en la especificidad del apareamiento entre bases. • Tiene las siguientes capacidades: – Reconoce las secuencias promotoras que marcan el inicio de la transcripción, para lo cual necesita la ayuda de otras proteínas: los factores de transcripción. – Recorre la hebra molde en sentido 3´- 5´. Lee la secuencia de sus bases y selecciona el ribonucleótido trifosfato, cuya base debe ser complementaria a la de la base del molde. – Cataliza la formación del enlace éster entre los nucleótidos. – Reconoce la secuencia de terminación de la transcripción.
- 15. Maduración postranscripcionalMaduración postranscripcional • La maduración del pre-ARNm consiste en un conjunto de modificaciones en ambosmodificaciones en ambos extremos de la moléculaextremos de la molécula y en la eliminacióneliminación de intronesde intrones.
- 16. Etapas de transcripciEtapas de transcripción del ARNmón del ARNm • El proceso de transcripción transcurre en tres etapas diferentes: IniciaciónIniciación, ElongaciónElongación y terminaciónterminación.
- 17. IniciaciIniciaciónón • Para asegurar que la enzima ARN polimerasa IIARN polimerasa II transcriba determinados genes en el momento preciso y a una velocidad adecuada, existen en los genes eucariotas tres clases de secuencias reguladoras: el promotorpromotor, las secuenciassecuencias potenciadoraspotenciadoras y las silenciadoras.silenciadoras.
- 18. • El promotorpromotor es la secuencia más próxima al inicio de la transcripción y está formada por la secuencia -25 TATATATA, junto con la secuencia -80 CAATCAAT y la secuencia -120 rica en bases GCGC • Las secuencias potenciadorassecuencias potenciadoras situadas entre -200 y - 10000. Desempaquetan la cromatina al disgregar los nucleosomas y consiguen desenrollar la doble vuelta del ADN para que resulte accesible la región promotora. • Las secuencias silenciadorassecuencias silenciadoras se encuentran intercaladas entre las activadoras y disminuyen la velocidad de la transcripción.
- 20. ElongaciElongaciónón • La enzima ARN polimerasa II recorre la hebra molde en sentido 3´-5´, mientras que la cadena del ARN transcrito primarioARN transcrito primario o pre-ARNmpre-ARNm continúa creciendo en la dirección 5´-3´ a razón de unos 30 nucleótidos por segundo.
- 21. TerminaciTerminaciónón • La enzima ARN polimerasa II reconoce la secuencia TTATTTTTATTT que determina el fin de la transcripción (puede continuar cientos de nucleótidos más allá de esta señal) y la separación de la cadena de pre-ARNm recién sintetizada de la hebra molde de ADN
- 22. Maduración postranscripcional • La maduración del pre-ARNm consiste en un conjunto de modificaciones en ambos extremosmodificaciones en ambos extremos de la moléculade la molécula y en la eliminación de loseliminación de los intronesintrones.
- 23. Adición en 5´de la caperuzaAdición en 5´de la caperuza de metil-guanosina trifosfatode metil-guanosina trifosfato • Poco después de iniciada la transcripción del gen, se le añade al extremo 5´ del pre-ARNm un resto de metil guanosina trifosfato. • Esta especie de caperuza protegeprotege del ataque de las nucleasas. • Además esta caperuza es reconocida por los ribosomas como lugar de inicio de la traducciónlugar de inicio de la traducción.
- 24. Adición en 3´de una cola deAdición en 3´de una cola de poli-adenina (poli-A)poli-adenina (poli-A) • Al final de la transcripción se añade, mediante la enzima poli-A polimerasa, una “cola de poli-A” al extremo 3´ del pre-ARNm. • Está formada por unos 150 a 200 ribonucleótidos de adenina. • Tiene un papel protector del ARNm e interviene en la vida media de esta molécula, pues cuanto menor es la cola de poli-A más rápidamente se degrada.
- 25. Mecanismo de corte deMecanismo de corte de intrones y pegado de exonesintrones y pegado de exones • El pre-ARNm contiene tanto secuencias llamadas exonesexones, que serán traducidas porque poseen información para la síntesis de la proteína correspondiente, como secuencias intercaladas llamadas intronesintrones, que no codifican para la síntesis de proteínas, y por tanto, deben ser eliminadas.
- 26. • La supresión de los intronessupresión de los intrones se realiza mediante un proceso de maduración en el núcleo del pre-ARNm que supone cortes entre los intrones y los exones, de manera que las secuencias intrónicas se enrollan en forma de lazos y se eliminan. • Las secuencias exónicas se empalmanexónicas se empalman y forman una molécula de ARNm funcionalARNm funcional que se exporta al citoplasma y contiene todos los nucleótidos necesarios para la síntesis de la proteína correspondiente.
- 28. Transcripción en procariotasTranscripción en procariotas • Es esencialmente igual que en eucariotas, aunque presenta algunas diferencias: • Requiere una cadena de ADN molde, ribonucleótidos y, a diferencia que las eucariota, una única enzima de ARN polimerasa. • También tiene iniciación, elongación y terminación. • Presenta un promotor (TATATT), pero no necesita descompactar la cromatina, ni existe la complejidad de los factores de transcripción, ni secuencias potenciadoras, ni inhibidoras. • La molécula de ARN recién transcrito no necesita un proceso de maduración, ya que los genes procariotas no poseen intrones ni exones.
- 29. TraducciTraducción:descodificación delón:descodificación del ARNmARNm • Es la etapa que sigue a la traducción mediante la cual se descodifica el mensaje genético que contiene el ARNm para que se sintetice una proteína. • El proceso de biosíntesis de proteínas se denomina traducción porque no es un proceso de copia a partir de un molde y en él se cambia de “idioma”: se pasa del idioma de los ácidos nucleicos, cuyo alfabeto posee cuatro letras (A, G, C y U) al idioma de las proteínas que posee un alfabeto de 20 letras (los 20 aminoácidos proteicos)
- 30. El cEl código o la clave genéticaódigo o la clave genética • Establece las correspondencias entre nucleótidos y aminoácidos, lo que permite traducir el idioma de los genes al de las proteínas; los aminoácidos están codificados por palabras de tres letras, que son tripletes de bases de ARNm, también llamados codones.
- 31. CaracterCaracterísticas del códigoísticas del código genéticogenético
- 32. 1.- El c1.- El código genético está degeneradoódigo genético está degenerado • Quiere decir que el diccionario es redundante: puesto que existen 61 codones que codifican 20 aminoácidos, algún aminoácido debe estar codificado por dos o más tripletes distintos, es decir, hay codones que significan lo mismo (codones sinónimos)
- 33. 2.- Es universal2.- Es universal • Es utilizado indistintamente por la práctica totalidad de los organismos conocidos (virus, bacterias, plantas, animales…)
- 34. 3.- No es ambiguo3.- No es ambiguo • Cada codón o triplete tiene siempre el mismo significado, es decir, especifica el mismo aminoácido.
- 35. 4.- Todos los tripletes tienen sentido4.- Todos los tripletes tienen sentido
- 36. 5.- Carece de solapamientos5.- Carece de solapamientos • Los tripletes se disponen en el ARNm uno a continuación de otro y no comparten ninguna base.
- 37. 6.- Es unidireccional6.- Es unidireccional • Se leen en el ARNm en sentido 5´-3´.
- 38. La funciLa función de intérprete de losón de intérprete de los ARNtARNt • Los ARNt son los verdaderos artífices de la traducción, pues se encargan de mantener lasmantener las correspondenciascorrespondencias entre los tripletes del ARNm y los aminoácidos. • En cada molécula de ARNtmolécula de ARNt se distinguen dos regiones distintas y separadas que permiten actuar como intérprete: la secuenciala secuencia CCA del extremo 3´CCA del extremo 3´ y el bucle delbucle del anticodónanticodón.
- 39. • Por un lado, cada ARNtARNt se une específicamente con único aminoácidoaminoácido determinado. Para ello la enzima aminoacil ARNtaminoacil ARNt sintetasasintetasa actúa como adaptador, ya que dispone de dos sitios activos altamente específicos: uno de ellos reconoce el aminoácidoaminoácido, el otro identifica determinadas secuencias de bases características de cada ARNt. • Esta enzima cataliza el enlace entre el grupo carboxilogrupo carboxilo (- COOH) del aminoácido con el grupo hidroxilogrupo hidroxilo (-OH) del ribonucleótido de adenina del extremo 3´(secuencia CCA).
- 40. Etapas de la traducciEtapas de la traducción enón en eucariotaseucariotas • La lectura por los ribosomas del mensaje genético transportado por el ARNm que conduce a la síntesis de una cadena peptídica es, en esencia, un proceso similar en procariotas y eucariotas y, en ambos casos, se puede considerar dividido en tres etapas sucesivas: iniciacióniniciación, elongaciónelongación de la cadena peptídica y terminaciónterminación.
- 41. IniciaciIniciación de la síntesis de la cadenaón de la síntesis de la cadena polipeptídicapolipeptídica • Comienza por el triplete iniciador AUG que esté más próximo a la “caperuza” de metil-guanosina-trifosfato del ARNm (extremo 5´) • El primer paso consiste en la construcción del complejo deconstrucción del complejo de iniciación 80 Siniciación 80 S, que está formado por un ribosomaribosoma unido al ARNmARNm y al ARNt iniciadorARNt iniciador cargado con el aminoácido metionina (ARNti Met ).
- 42. La formación del complejo de iniciación se lleva a cabo en varias fases sucesivas: • 1.-1.- Determinados factores de iniciacifactores de iniciaciónón FIFI y la energía que suministra el GTPGTP provocan la unión de la subunidad pequeña del ribosoma con el ARNt iniciador cargado con el aminoácido metionina (ARNti Met ).
- 43. • 2.-2.- Otros factores de iniciación facilitan que la subunidad menor del ribosoma, unida al ARNti Met , reconozca la “caperuza” de metil-guanosina-trifosfato y se una con el ARNm en el extremo 5´.
- 44. • 3.-3.- La subunidad pequeña del ribosoma se desplaza por el ribosoma en sentido 5´- 3´sentido 5´- 3´, con la energía aportada por el ATPATP, y rastrea la secuencia del ARNm hasta encontrar el primer codón de iniciación AUG. En este momento se produce la unión con el ARNti Met , el cual posee el anticodón UAC
- 45. • 4.-4.- Al encajar el ARNti Met exactamente en el codón de iniciación AUG, se liberan los FIFI y dejan paso a la subunidad mayor del ribosoma, que se acopla con la subunidad pequeña, el ARNm y el ARNti Met , para formar el complejo de iniciación 80 Scomplejo de iniciación 80 S completo y funcionalcompleto y funcional. En la subunidad mayor del ribosoma se localizan tres hendiduras o sitios de fijación AA, PP y EE:
- 47. ElongaciElongación de la cadenaón de la cadena peptídicapeptídica • Es el proceso catalizado por el complejo enzimático peptidil transferasapeptidil transferasa mediante el cual los sucesivos aminoácidos que se van añadiendo a la cadena peptídica, en el seno del ribosoma, quedan unidos mediante un enlace peptídico. • La incorporación de cada aminoácido se lleva a cabo mediante un proceso de elongación que consta de trestres fasesfases; cuando finaliza la tercera, comienza un nuevo proceso de elongación al incorporarse un nuevo aminoácido transportado por su correspondiente ARNt, y así sucesivamente.
- 48. Primera fase:Primera fase: entrada del ARNt-aminoentrada del ARNt-aminoácidoácido • El sitio P está ocupado inicialmente por el ARNti Met , y los sitios A y E están vacios. En nuestro ejemplo el triplete siguiente al de iniciación es el codón ACU que es la señal para que se aloje en el sitio A el ARNtThr , cuyo anticodón es UGA. Interviene el factor de elongación (FE-1FE-1) y se utiliza la energía suministrada por el GTP
- 49. Segunda fase:Segunda fase: formaciformación del enlace peptídicoón del enlace peptídico • El complejo enzimático peptidil transferasa cataliza la siguiente reacción: la metionina, unida con su grupo carboxilo (-COOH) al ARNti, rompe su enlace y se vuelve a asociar mediante un enlace peptídico con el grupo amino (-NH2) de la treonina, que continúa enlazada a su ARNt. El resultado es la formación de un dipéptido (Met-Thr) unido al ARNt de la treonina y alojado provisionalmente en el sitio A.
- 50. Tercera fase:Tercera fase: TraslocaciTraslocaciónón • Interviene un segundo factor de elongación (FE-2)(FE-2) que, utilizando la energía suministrada por el GTPGTP, obliga al ribosoma a desplazarse exactamente tres nucleótidos a lo largo del ARNm, en sentido 5´- 3´: primero se traloca y avanza la subunidad grande del ribosoma, quedando un nuevo codón en el sitio A libre, y luego lo hace la subunidad pequeña. Este desplazamiento trasloca todo el complejo peptidil-ARNt-ARNm del sitio A al sitio P.
- 51. • El sitio PEl sitio P queda ocupado por el péptido en formación unido al ARNt (en nuestro ejemplo, Met-Thr-ARNt) • El sitio AEl sitio A queda vacante y dispuesto a recibir a otro ARNt cargado con su correspondiente aminoácido (en nuestro caso ARNtPhe ) • El sitio EEl sitio E queda ocupado con el ARNti correspondiente a la metionina liberada de su carga
- 52. Fases sucesivasFases sucesivas • La entrada de un tercer ARNt cargado con su correspondiente aminoácido( Phe) provoca la salida del ARNti del sitio E y se repiten las tres fases anteriores. Y así transcurre el proceso de elongación ( a un ritmo de unos 3 a 5 aminoácidos por segundo)
- 54. TerminaciTerminación de la síntesis de la cadenaón de la síntesis de la cadena peptídicapeptídica • La sintesis se detiene en el mometo de producirse la última traslocación del ribosoma, aparece en el sitio A uno de los tres codones de terminacióncodones de terminación del ARNm (UUA, UAG o UGA) • En este momento, un factor proteico de terminación (RF)factor proteico de terminación (RF) se une al codón de terminación (en nuestro caso UAG) e impide que algún aminoacil-ARNt se aloje en el sitio A. El complejo enzimático peptidil transferasa se ve obligada a catalizar la transferencia de la cadena peptídica, no a otro aminoácido, sino a una molécula de agua, lo que provoca la hidrólisis del enlace entre el péptido recién sintetizado y el ARNt.
- 57. • Maduración postraduccinal de las proteínas. • Plegamiento postraduccional de las proteínas: chaperonas moleculares
- 58. REPLICACIREPLICACIÓN DEL ADNÓN DEL ADN
- 59. REPLICACIREPLICACIÓN DE LA DOBLE HÉLICEÓN DE LA DOBLE HÉLICE • Cada hebra se separa y actúa de moldemolde o patrón para la síntesis de una nueva cadena que posee una secuencia desecuencia de bases complementariabases complementaria. • Se dice que se ajusta a un modelo semiconservativosemiconservativo, pues en la doble hélice de cada célula hija se conserva una cadena original de la célula madre; la otra cadena se sintetiza de nuevo.
- 60. ReplicaciReplicación del ADN en bacteriasón del ADN en bacterias • Comienza cuando se forma una burbuja de replicación, que se extiende y da lugar a dos horquillas de replicación que se desplazan en sentidos opuestos hasta que se encuentran en el punto de terminación.
- 61. • Comienza en un punto y se desarrolla en tres etapas:Comienza en un punto y se desarrolla en tres etapas: • Apertura y desenrollamiento de la doble hApertura y desenrollamiento de la doble héliceélice • Síntesis de dos nuevas cadenas de ADNSíntesis de dos nuevas cadenas de ADN • Corrección de errores.Corrección de errores.
- 62. 1.- Apertura y desenrollamiento de la doble h1.- Apertura y desenrollamiento de la doble héliceélice • La separación de las cadenas comienza en un punto concreto del cromosoma denominado oriC, rico en secuencias GATC, que marca el origen de replicación.
- 63. • A partir del origen de replicación se forman unas estructuras, conocidas como burbujas de replicaciónburbujas de replicación, que se extienden a lo largo del cromosoma, en sentidos contrarios, y dan lugar a dos horquillas de replicaciónhorquillas de replicación (por su forma en Y), donde las dos hebras del ADN parental están separadas y actúan como moldes para la síntesis de dos nuevas cadenas de ADN: por esa razón se dice que la replicación es bidireccionalreplicación es bidireccional.
- 64. • Para que la doble hélice se desenrolle sin que las hebras circulares acaben enredándose en un ovillo, interviene un conjunto de proteínas y enzimas que, junto con las enzimas ADN polimerasas, constituyen un aparato molecular de replicación denominado replisomareplisoma:
- 65. • En primer lugar actúan las enzimas helicasashelicasas que facilitan el desenrollamiento de la doble hélice. • Para eliminar las tensiones generadas por la torsión intervienen las girasasgirasas y las topoisomerasastopoisomerasas. • Luego, las proteínas SSBPproteínas SSBP se unen a la hebra molde del ADN para estabilizarlas y que no se vuelvan a enrollar.
- 66. 2.- S2.- Síntesis de dos nuevas cadenas de ADNíntesis de dos nuevas cadenas de ADN • Una vez formada la burbuja de replicación, ya pueden actuar las enzimas ADNADN polimerasaspolimerasas que leen la secuencia de cada una de las cadenas y elaboran dos copias con secuencias complementarias • La ADN polimerasa IIIADN polimerasa III es la que lleva a cabo la mayor parte del proceso
- 67. • Recorre las hebras moldeRecorre las hebras molde y seleccionaselecciona en cada momento el desoxirribonucleótido trifosfato, cuya base debe ser complementaria a la base del molde. • Si el nucleótido seleccionado es el complementario, cataliza sucataliza su hidrólisishidrólisis, separando un resto pirofosfato(PP) e incorporandoincorporando el nucleótido monofoafato a la cadena de ADNel nucleótido monofoafato a la cadena de ADN en formación mediante enlaces fosfodiéster, para el que utiliza la energía desprendida en la hidrólisis.
- 69. Problemas que debe resolver la enzima ADN polimerasa IIIProblemas que debe resolver la enzima ADN polimerasa III a).- Inicio de la sa).- Inicio de la síntesis.íntesis. • Es incapaz de iniciar por sí sola la síntesis pues necesitanecesita un grupo hidroxilo libreun grupo hidroxilo libre (-OH) • Necesita la colaboración de una enzima de ARNARN polimerasapolimerasa (primasa) que sintetiza un ARN cebadorARN cebador (primer) que proporciona extremos hidroxilo libres sobre los que adicionar nuevos nucleótidos.
- 70. b).- Sentido de lectura de la hebra moldeb).- Sentido de lectura de la hebra molde • La ADN polimerasa IIIADN polimerasa III solo ”sabe leer” las secuencias de las hebras en sentido 33´- 5´´- 5´, mientras que las nuevas cadenas se sintetizan y crecen en sentido 5´- 3´5´- 3´. Por lo tanto, de las dos hebras molde del ADN, la que está orientada en sentido 3´- 5´3´- 5´ es copiada de manera continua y la nueva réplica recibe el nombre de hebra conductorahebra conductora o lider.
- 71. • Sin embargo la otra hebra molde, al ser antiparalelaantiparalela tiene sentido 55´- 3´´- 3´ y no puede ser leída. • Este problemas se soluciona abriendo porciones de la hebra molde suficientemente grandes que permitan a la enzima ir retrocediendo para poder leer en el sentido adecuado y así sintetizar pequeños fragmentos de ADN, llamados fragmentosfragmentos de Okazakide Okazaki, que crecen en el sentido 5´- 3´5´- 3´ y, más tarde, se unen para formar la otra réplica que recibe el nombre de hebrahebra retardadaretardada.
- 72. Modelo de horquilla de replicaciModelo de horquilla de replicación enón en Escherichia coli • Cada fragmento comienza cuando un ARN polimerasaARN polimerasa, llamada primasa, fabrica una corta cadena de ARN cebadorARN cebador. • Esta cadena es alargada por la ADN polimerasa IIIADN polimerasa III, que sintetiza un pequeño trozo de ADN hasta completar un fragmento de Okazakifragmento de Okazaki. • Más tarde actúa la ADN polimerasa IADN polimerasa I, que elimina el ARN cebador de cada fragmento. El hueco que queda se rellena por la ADN polimerasa I, que sustituye el ARN cebador por ADN, con una secuencia complementaria a la del molde • Por último, interviene una enzima ADN ligasaADN ligasa para unir los diferentes fragmentos hasta formar la hebra retardada.
- 73. • La hebra conductoraLa hebra conductora se sintetiza con mayor rapidez debido a que las enzimas primasa, y ADN polimerasas I y III actúan una sola vez, al principio de la réplica • La hebra retardadaLa hebra retardada se sintetiza de forma discontínua, mediante un proceso que requiere la colaboración de varias enzimas.
- 74. 3.- Correcci3.- Corrección de erroresón de errores • a).- Actividad autocorrectora de la ADN polimerasa: correccia).- Actividad autocorrectora de la ADN polimerasa: correcciónón de pruebas.de pruebas. • Las ADN polimerasa I y III además de polimerizar, son autocorrectorasautocorrectoras, ya que realizan una nueva lectura: después de cada proceso de polimerización, la ADN polimerasa “mira” hacia atrás antes de incorporar el nucleótido siguiente y, si detecta un error, elimina el último nucleótido y vuelve a introducir el adecuado. • En E. Coli se comete un error de apareamiento por cada 101066 nucleótidos adicionados. La posterior actividad autocorrectora reduce el error de apareamiento a uno por cada 101088 nucleótidos adicionados.
- 75. b) Correccib) Corrección postreplicativa: reparación del malón postreplicativa: reparación del mal apareamiento.apareamiento. • Para aumentar todavía más la precisión de la replicación, existe una maquinaria enzimática que corrige los posibles errores cometidos por las ADN polimerasa I y III, que se denomina corrección postreplicativacorrección postreplicativa. • Incrementa unas 100 veces la exactitud de la réplica con lo que tenemos un error por cada 10101010 pares de bases. • Esta corrección se realiza por medio de enzimas correctoras, agrupadas en el replisomareplisoma, que detectandetectan el nucleótido mal emparejado, lo eliminaneliminan y regeneranregeneran la secuencia correcta.
- 76. • Para detectar el nucleótido mal apareado, el aparato de corrección debe reconocer la cadena recién sintetizada, donde se encuentra el errorerror, y diferenciarla de la cadena molde.
- 77. • La eliminación se realiza mediante una enzima endonucleasaendonucleasa que corta el segmento en el lugar donde se encuentra el nucleótido mal apareado.
- 78. • Por último, la secuencia correcta se regenera cuando la ADN polimerasa IADN polimerasa I rellena el hueco utilizado como molde la cadena parental complementaria: entonces el extremo 3´ del nuevo segmento se une con el segmento 5´ de la hebra réplica por acción de una enzima ADN ligasaADN ligasa
- 79. ReplicaciReplicación del ADN en células eucariotasón del ADN en células eucariotas • Transcurre en la fase S del ciclo celular de modo similar al descrito para los procariotas: – Es semiconservativasemiconservativa y bidireccionalbidireccional – La hebra conductorahebra conductora se sintetiza de manera continuacontinua y la retardadaretardada de forma discontinuadiscontinua, en forma de fragmentosfragmentos de Okazakide Okazaki que luego se unen; – También se requiere un ARN cebadorARN cebador para que inicie la replicación del ADN. Sin embargo presenta ciertas características peculiares:
- 80. • Intervienen, al menos, cinco tipos de ADN polimerasa: • αα ( sintetiza el ARN cebador y la hélice retardada), • ββ (une fragmentos de Okazaki), • γγ (replica el ADN mitocondrial), • δδ (sintetiza la hebra conductora) • εε (polimeriza los segmentos de Okazaki)
- 81. • El ADN se encuentra asociado con los octoctómeros de histonasómeros de histonas, en forma de nucleosomasnucleosomas, por lo que, además de replicarse el ADN, deban duplicarse también las histonas.
- 82. • Consta de unos 3 x 103 x 1099 pares de basespares de bases. Si se replicase al ritmo que en E. Coli (30 minutos), tardaría 30.000 minutos.. • Para evitar esto en cada cromosoma se forman numerosas burbujas de replicaciónburbujas de replicación. En el genoma humano pueden existir unos 30.000 puntos de inicio.
- 83. Mutaciones y el cáncerMutaciones y el cáncer • Las células que forman parte de los tejidos se caracterizan por estar sujetas al control general del organismo, que indica cuándo deben dividirse y cuándo debe finalizar el crecimiento del tejido. • Las mutaciones carcinógenas desencadenan una serie de procesos de transformación que convierten a las células en cancerígenas o tumorales. • Estas células se dividen de manera incontrolada y se diseminan por todo el organismo, invadiendo los tejidos sanos.
- 84. • El tumor benignotumor benigno, como las verrugas, permanece confinado en el lugar donde apareció y no se propaga a otros tejidos. • El tumor malignotumor maligno o cáncer es capaz de invadir tejidos adyacentes, propagarse y dar lugar a metástasis. • Cáncer o neoplasiaCáncer o neoplasia son términos que engloban a un conjunto de enfermedades que tienen en común una proliferación desordenada de las células que forman un tumor con capacidad de invadir los tejidos adyacentes y de propagarse por todo el organismo • Las etapas del desarrollo de un cáncer son:
- 85. InicioInicio • Una célula hereda un oncogén o lo adquiere a lo largo de su vida, y transmite la predisposición a dividirse exageradamente a todo a todo el clon de células descendentes.
- 86. HiperplasiaHiperplasia • La célula mutada y su clon, aparentemente normal, prolifera en exceso.
- 87. DisplasiaDisplasia • Una célula mutada adquiere una segunda mutación que modifica su aspecto, se divide exageradamente y transmite esta carácter a su descendencia
- 88. NeoplasiaNeoplasia • Una de estas células adquiere una tercera mutación que modifica aún más su morfología y su carácter proliferativo, hasta que alguna de las descendientes del clon celular ha acumulado las mutaciones necesarias para transformarse en un cáncer localizado.
- 89. MetástasisMetástasis • Nuevas mutaciones liberan a las células cancerosas de las restricciones impuestas por los sistemas de regulación, proliferan sin control y llegan a invadir y colonizar otros tejidos: El cáncer se hace invasivo y se vasculariza.
