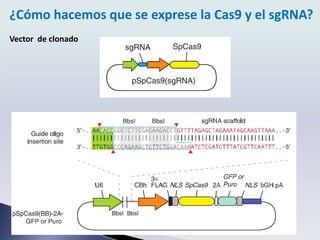
El sistema CRISPR-Cas9 permite editar genomas de forma altamente específica y eficiente mediante el uso de una enzima Cas9 y un ARN guía para cortar el ADN en una secuencia diana. Esto puede usarse para eliminar, reemplazar o modificar genes, creando nuevas posibilidades para la ingeniería genética y la cura de enfermedades.