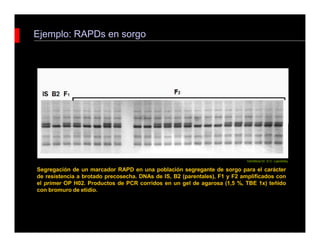
Este documento describe diferentes tipos de marcadores moleculares, incluyendo marcadores bioquímicos como isoenzimas y proteínas de reserva, y marcadores genotípicos como RFLP, RAPD y AFLP. Explica cómo estos marcadores permiten detectar polimorfismos a nivel genético que pueden usarse para mapeo genético y otros estudios.