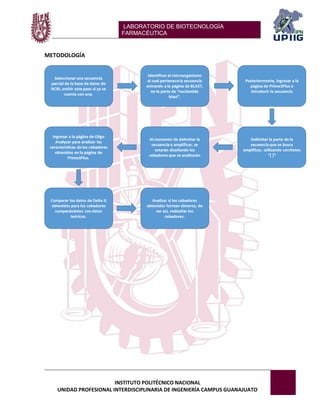
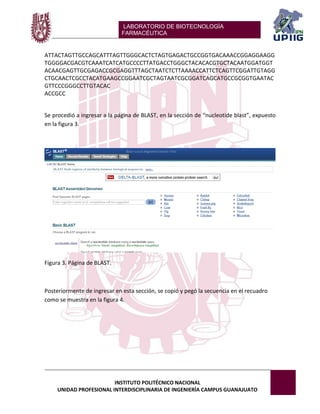
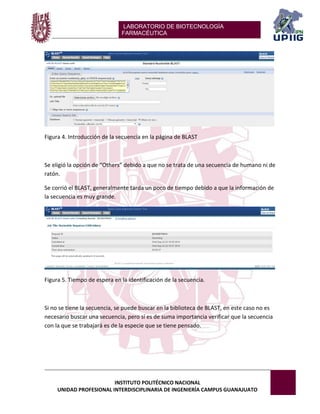
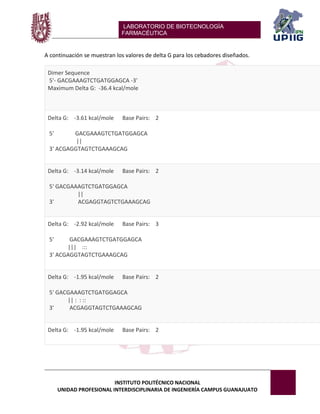
Este documento describe un experimento de bioinformática que involucra el análisis de una secuencia de ADN de la bacteria Lactobacillus sakei utilizando la herramienta BLAST. Se seleccionó una parte de la secuencia de L. sakei y se cargó en BLAST para identificar de qué organismo proviene y obtener información relevante sobre cómo fue obtenida y para qué propósito. Los resultados muestran que la secuencia corresponde efectivamente a L. sakei y proporcionan detalles sobre su diversidad genética y uso de cebadores de PCR.









![LABORATORIO DE BIOTECNOLOGÍA
FARMACÉUTICA
INSTITUTO POLITÉCNICO NACIONAL
UNIDAD PROFESIONAL INTERDISCIPLINARIA DE INGENIERÍA CAMPUS GUANAJUATO
Query 1321 AACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATA 1380 |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||| Sbjct 1321 AACTCGCCTACATGAAGCCGGAATCGCTAGTAATCGCGGATCAGCATGCCGCGGTGAATA 1380 Query 1381 CGTTCCCGGGCCTTGTACACACCGCC 1406 |||||||||||||||||||||||||| Sbjct 1381 CGTTCCCGGGCCTTGTACACACCGCC 1406
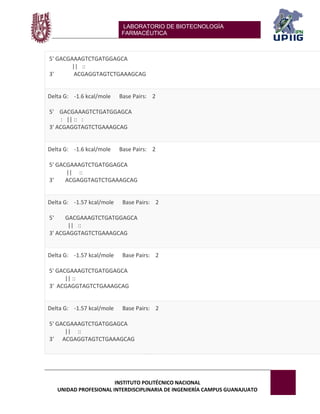
Ya que contamos con todas las alineaciones de la secuencia parcial de ADN de esta bacteria, procedemos a introducir la secuencia en la página de Prime3plus.
Esta página nos sirve para seleccionar y diseñar nuestros primers u oligonucleótidos.
Figura 7. Página de Primer3Plus.
Como se observa en la Figura 7, primero se debe seleccionar la parte de la secuencia que se busca amplificar por medio de la técnica de PCR, para seleccionar esta sección debemos utilizar los corchetes “[ ]”, que se muestran en la parte inferior de la Figura 7, y automáticamente al seleccionar nuestra región para amplificar, se seleccionarán los primers.](https://image.slidesharecdn.com/reporteprctica3-141111221830-conversion-gate02/85/Reporte-practica-3-13-320.jpg)





![LABORATORIO DE BIOTECNOLOGÍA
FARMACÉUTICA
INSTITUTO POLITÉCNICO NACIONAL
UNIDAD PROFESIONAL INTERDISCIPLINARIA DE INGENIERÍA CAMPUS GUANAJUATO
REFERENCIAS
[1] PHOEBE CHEN, Yi-Ping. Bioinformatics Technologies. Alemania: Springer-Verlag Berlin Heidelberg, 2005. 396p. ISBN 3-540-20873-9.
[2] XION, Jin. Essential Bioinformatics. Estados Unidos de América: Cambridge University Press, 2006. 331p. ISBN 978-0-511-16815-4.
[3] HARJINDER S, Gill y PRAKASH C, Rao. Data Warehousing. La Integracion de Informacion para la Mejor Toma de Decisiones. México: Prentice Hall, 1996. 382p. ISBN 968-880-792-3.
[4] BioStar models of clinical and genomic data for biomedical data warehouse design [en línea]. WANG, Liangjiang; RAMANATHAN, Murali y ZHANG, Aidong.State University of New York at Buffalo: New York, Estados Unidos de América, 2005 - [citado el 30 de marzo de 2011]. Disponible desde Internet en: http://www.cse.buffalo.edu/DBGROUP/bioinformatics/papers/ijbra05.pdf
[5] PHOEBE CHEN, Yi-Ping. Bioinformatics Technologies. Alemania: Springer-Verlag Berlin Heidelberg, 2005. 396p. ISBN 3-540-20873-9.
[6] GEER, Renata C. y SAYERS, Eric W. Entrez: Making use of its power. En: Briefings in Bioinformatics. vol. 4, no. 2. Junio, 2003. p. 179.
[7] FU, Limin. Knowledge Discovery Based on Neural Networks. En: Communications of the ACM (CACM). vol. 42, Issue: 11, Noviembre 1999. p. 47-50.
[8] UBERBACHER, Edward y Mural, Richard. Locating Protein Coding Regions in Human DNA Sequences Using a Multiple Sensor-Neural Network Approach. En: Proceedings of the National Academy of Sciences of United States of America. vol. 88, Diciembre de 1991. p. 11261-11265.](https://image.slidesharecdn.com/reporteprctica3-141111221830-conversion-gate02/85/Reporte-practica-3-21-320.jpg)