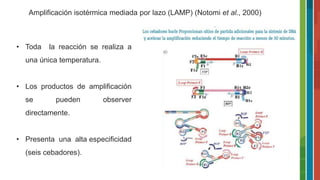
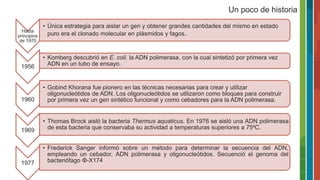
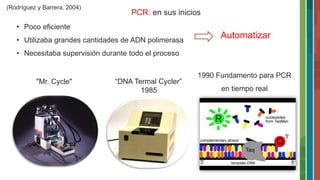
El documento detalla la historia y evolución de la reacción en cadena de la polimerasa (PCR) desde sus inicios en la década de 1970 hasta las técnicas avanzadas de PCR en tiempo real. Se describen los componentes necesarios, tipos de PCR y sus aplicaciones en microbiología y diagnóstico. Además, se mencionan las ventajas y desventajas de la técnica, así como variaciones como la PCR de extensión solapada y la PCR múltiplex.





![Empleo de adyuvantes: aumentan la especificidad y fidelidad.
• Dimetilsulfóxido: disminuye la estructura secundaria del ADN.
• Tween 20, Tritón X-100: estabilizan la enzima.
• Polietilenglicol, glicerol, formamida o seroalbúmina bovina…
Magnesio
Manganeso
0.5-2.5mM
Magnesio: bajo rendimiento.
Magnesio. Amplificaciones inespecíficas.
dATP
dCTP
dGTP
dTTP
0.2 a 1 mM
[ ] disminuyen la fidelidad de la
polimerasa e incluso pueden inhibir su
actividad
[]Magnesio=0.5-1mMx[]dNTPs
(Garrido, 2013)](https://image.slidesharecdn.com/pcr-181116161712/85/PCR-8-320.jpg)