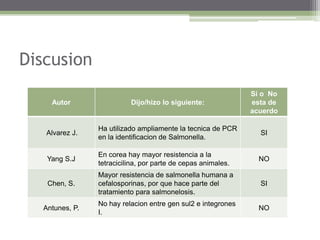
Este documento resume un estudio sobre la resistencia a antibióticos en cepas de Salmonella Typhimurium aisladas de humanos y animales en Malasia. Se caracterizaron 47 cepas mediante antibiogramas, detección de genes de resistencia, integrones y PFGE. La mayoría de cepas fueron resistentes a múltiples antibióticos y portaban genes y estructuras genéticas asociadas a resistencia. El estudio proporciona información sobre los mecanismos de resistencia en estas cepas y su importancia para la salud pública.