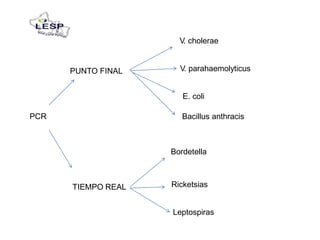
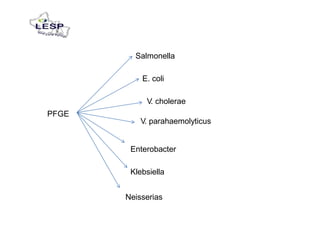
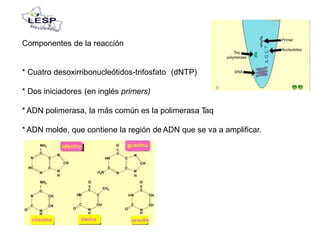
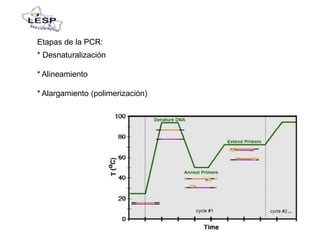
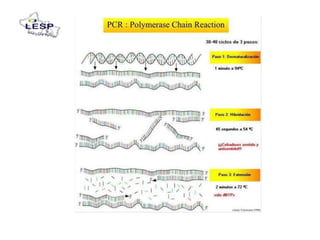
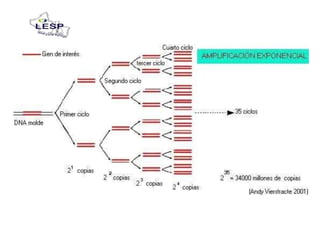
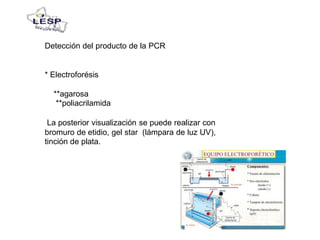
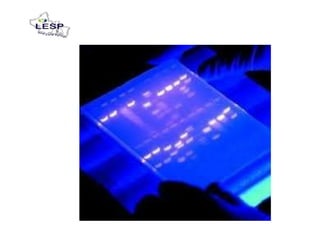
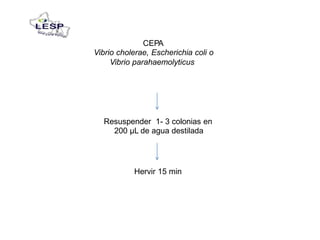
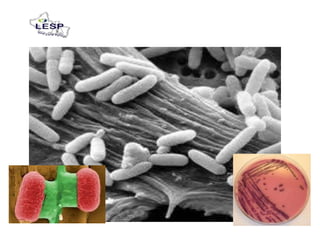
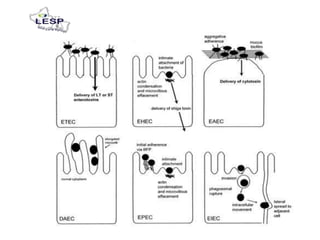
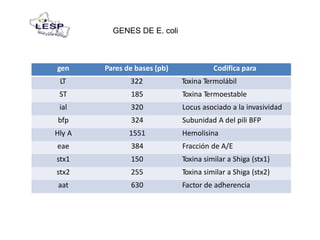
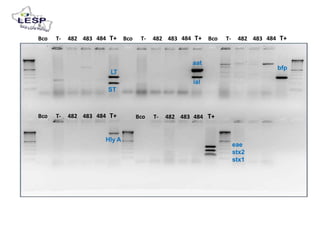
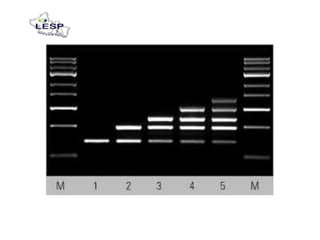
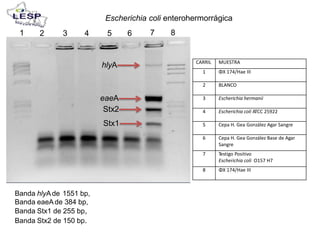
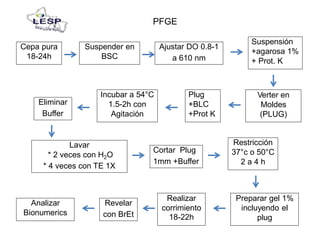
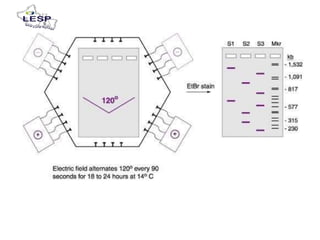
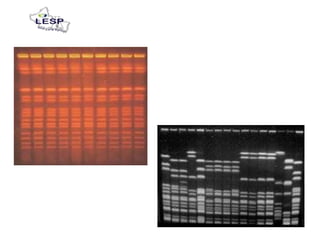
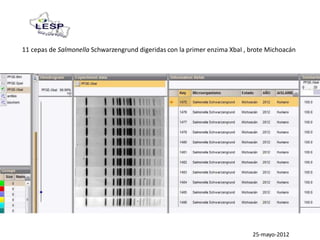
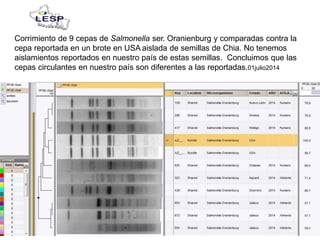
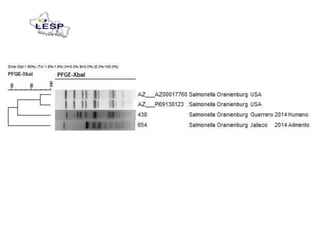
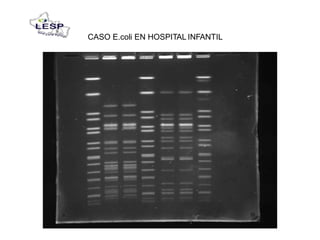
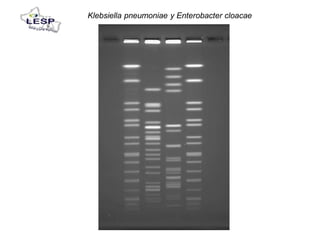
Este documento describe las pruebas moleculares utilizadas para diagnosticar bacterias como Vibrio cholerae, Vibrio parahaemolyticus, Salmonella spp. y E. coli. Brevemente describe la reacción en cadena de la polimerasa (PCR), la electroforesis en gel de campo pulsante (PFGE) y su uso para la epidemiología molecular y detección de brotes.