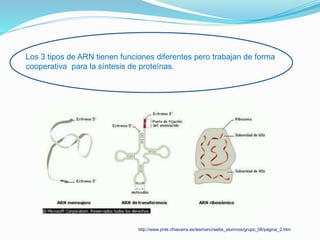
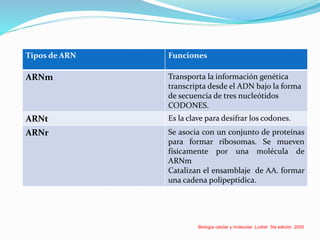
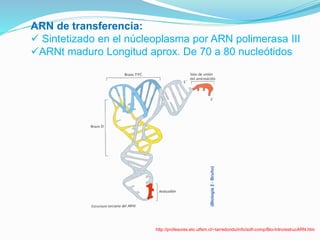
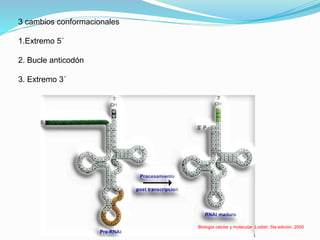
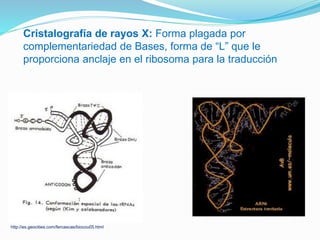
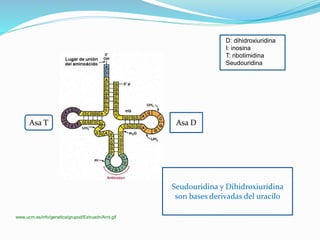
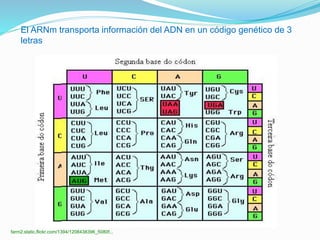
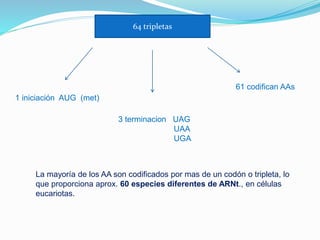
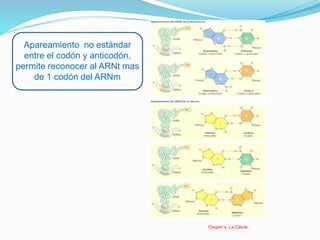
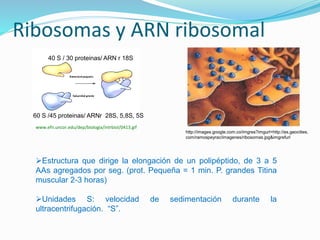
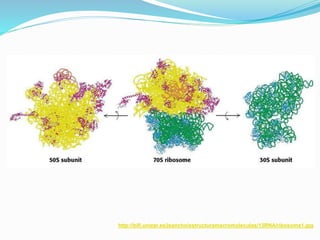
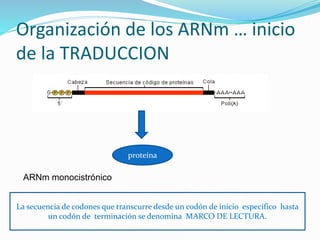
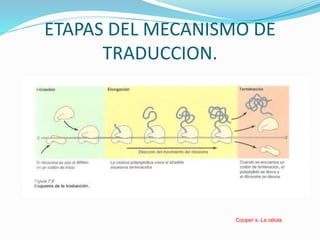
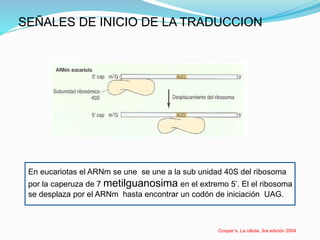
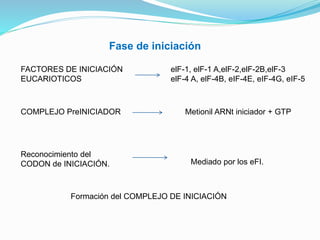
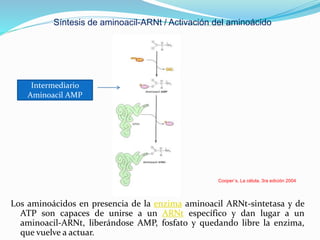
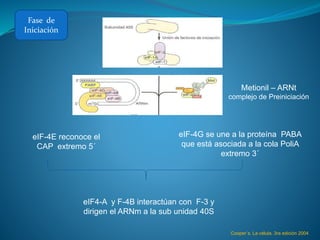
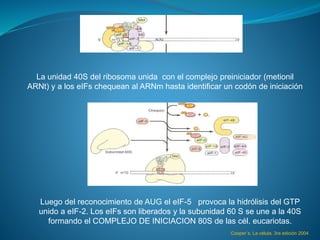
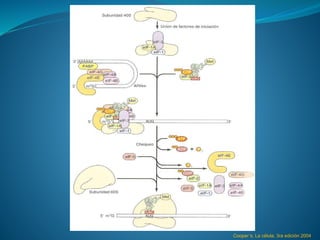
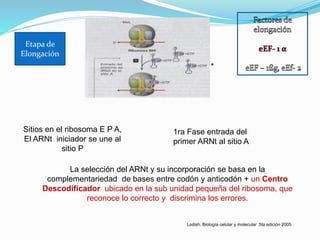
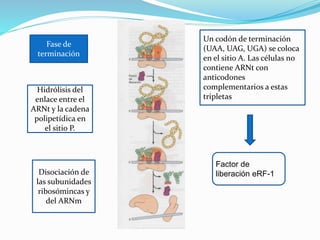
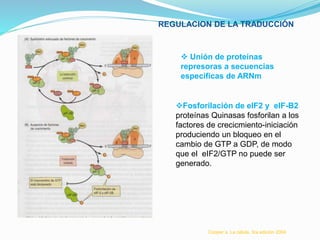
Este documento describe el proceso de síntesis de proteínas o traducción en células eucariotas. Los tres tipos de ARN (ARNm, ARNt y ARNr) trabajan juntos para ordenar los aminoácidos en una cadena polipeptídica. El ARNm transporta la información genética del ADN al ribosoma mediante codones, mientras que el ARNt reconoce los codones y el ARNr forma parte del ribosoma, donde se cataliza la unión de los aminoácidos. La traducción implica las fases de iniciación, elongación