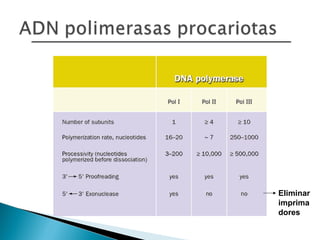
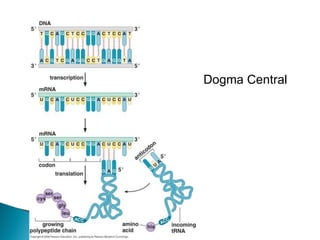
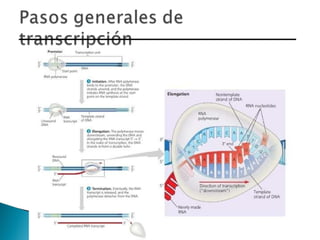
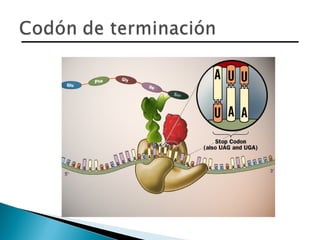
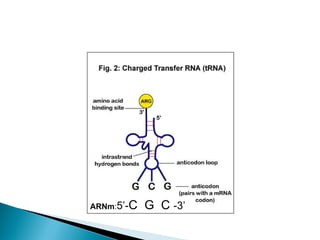
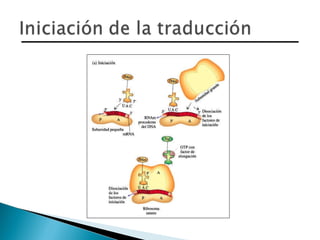
El documento aborda la estructura del ADN y sus componentes básicos, los nucleótidos, así como el modelo de doble hélice propuesto por Watson y Crick en 1953. También se discuten los procesos de replicación, transcripción y traducción, explicando cómo se codifican las proteínas a partir de la secuencia de nucleótidos en el ADN. Se menciona la importancia de las polimerasas y las diferencias entre la regulación genética en procariotas y eucariotas.