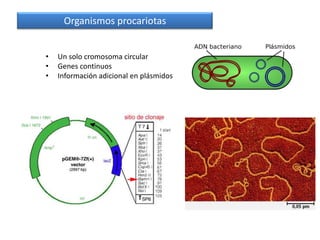
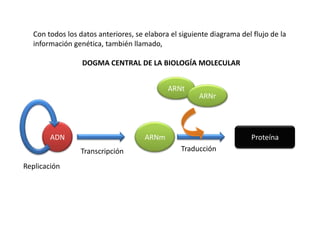
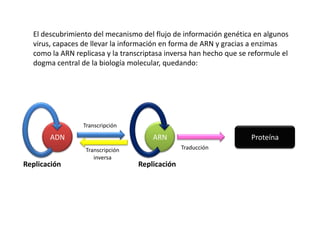
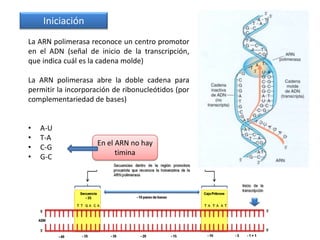
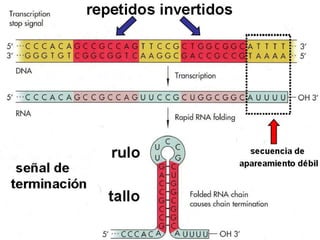
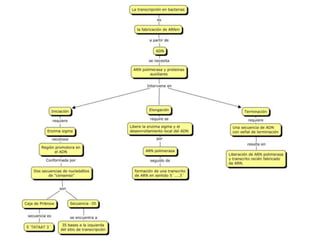
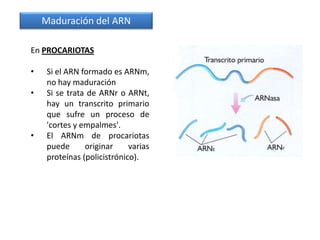
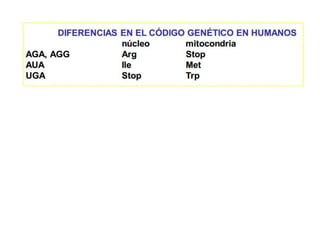
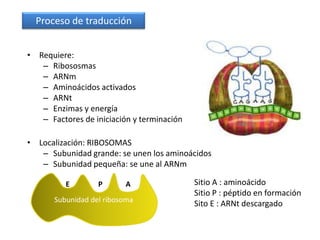
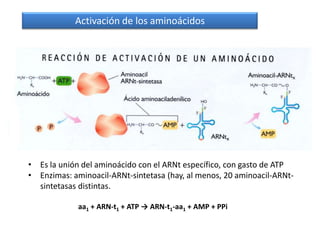
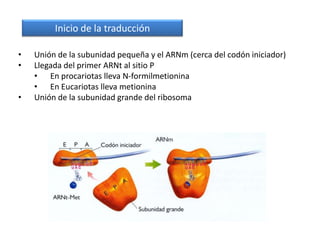
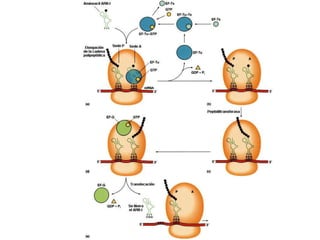
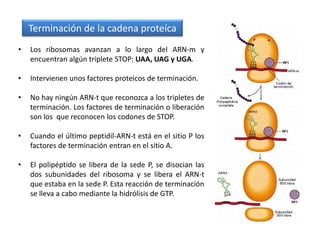
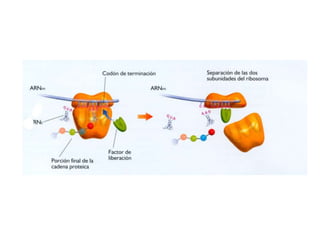
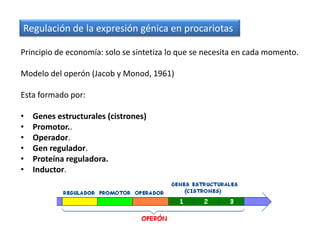
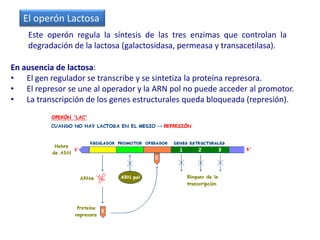
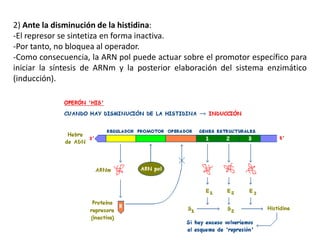
El documento describe el descubrimiento del flujo de la información genética desde el ADN hasta la síntesis de proteínas. Se detalla el proceso de transcripción del ADN en ARNm, y la traducción del ARNm en proteínas por los ribosomas. El código genético fue descifrado experimentalmente, determinándose que está formado por tripletes de nucleótidos (codones) que especifican los aminoácidos.