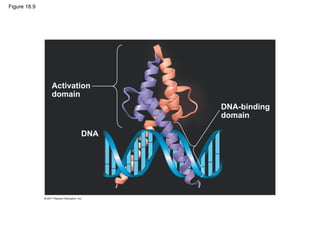
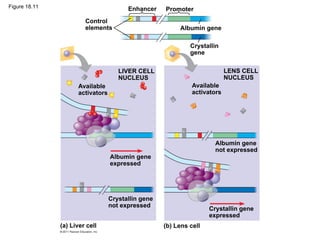
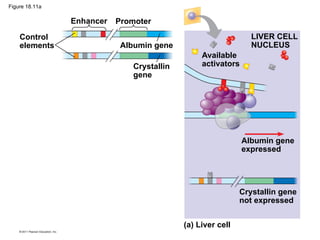
Este documento describe los mecanismos de regulación de la expresión génica en organismos eucariotas. La expresión génica está regulada a través de modificaciones en la cromatina, procesamiento del RNA, iniciación de la traducción y procesamiento de proteínas. Factores como la acetilación de histonas, metilación del DNA y procesamiento alternativo del RNA influyen en la expresión de genes específicos en diferentes tipos celulares.