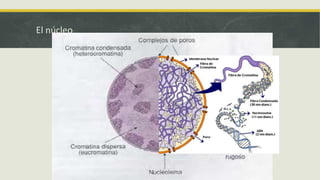
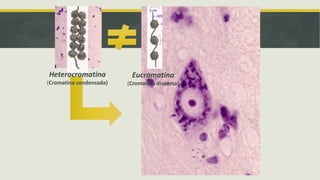
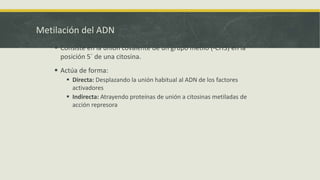
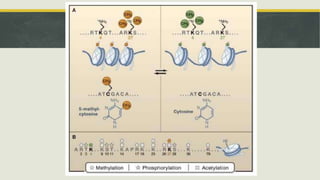
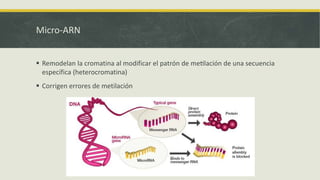
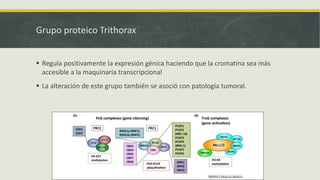
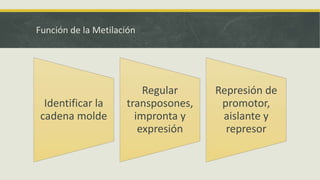
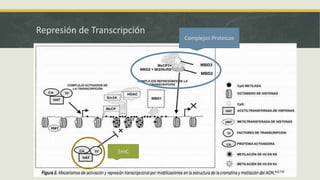
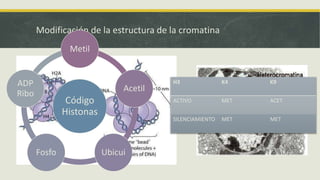
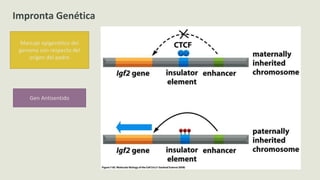
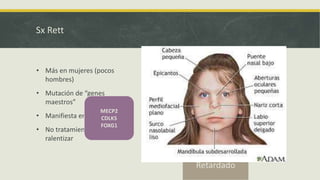
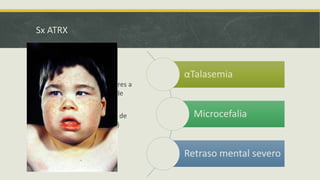
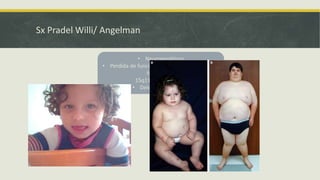
El documento describe los principales mecanismos epigenéticos que regulan la expresión génica, incluyendo la metilación del ADN, las modificaciones post-traduccionales de las histonas, el silenciamiento mediado por ARN no codificante y el remodelado de la cromatina dependiente de ATP. También explica cómo alteraciones en estos mecanismos epigenéticos pueden dar lugar a síndromes y enfermedades como el síndrome ICF, el síndrome de Rett, el síndrome de ATRX y el sínd