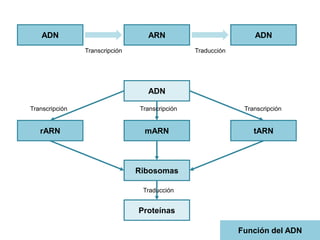
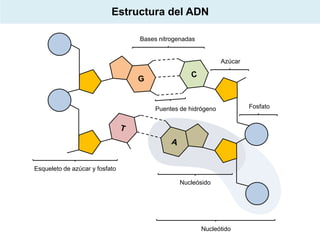
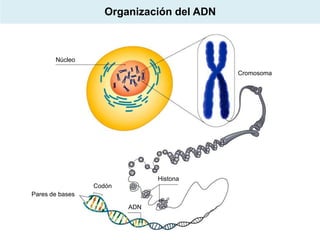
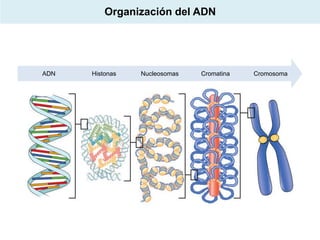
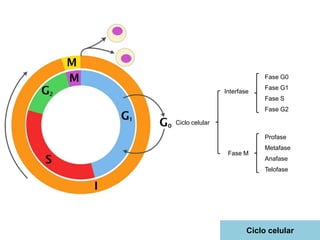
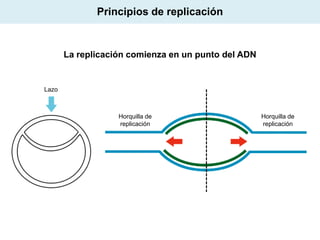
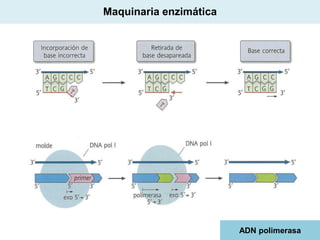
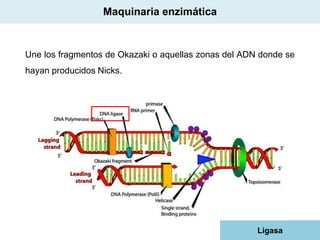
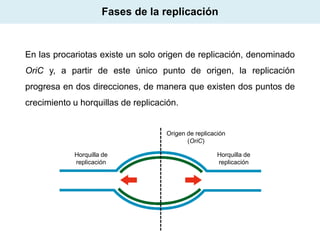
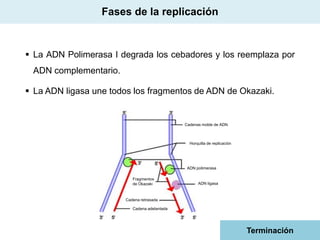
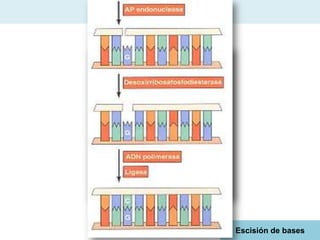
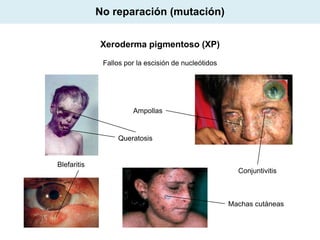
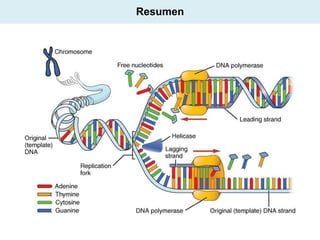
El documento aborda la replicación del ADN, un proceso esencial para la herencia y la síntesis de proteínas, describiendo su estructura, funciones, y los principales mecanismos enzimáticos involucrados. Se explica la naturaleza semiconservadora de la replicación, así como las fases del ciclo celular y los tipos de daños y reparaciones del ADN. También se detallan las diferencias en la replicación entre procariotas y eucariotas, haciendo hincapié en los fragmentos de Okazaki y las proteínas esenciales para el proceso.