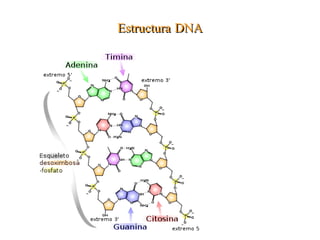
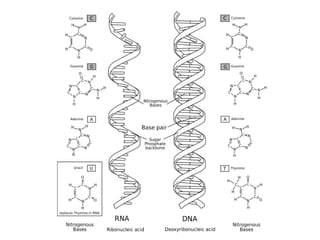
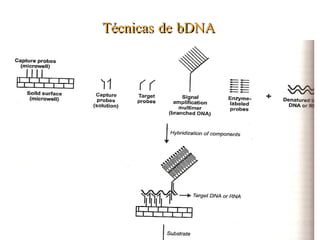
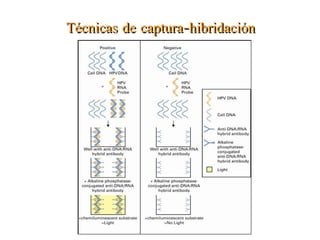
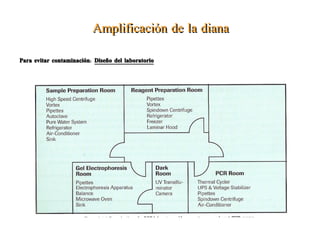
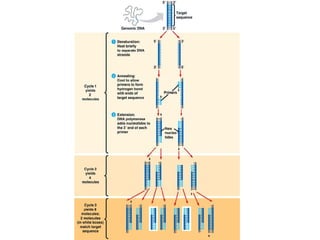
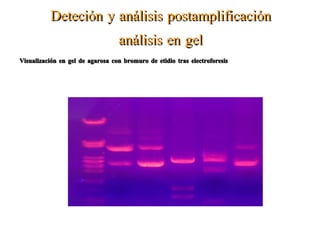
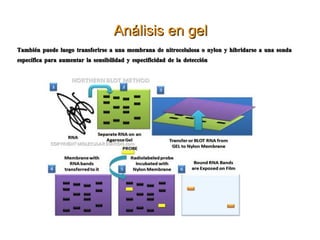
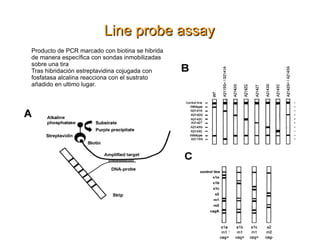
El documento describe varios métodos para la extracción y detección de ADN, incluyendo la extracción de ADN de tejido, detección mediante sondas de hibridación, técnicas de amplificación como PCR y RT-PCR, y métodos automatizados para la extracción y detección de ADN.