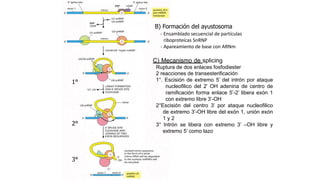
El documento trata sobre la transcripción de ADN y los procesos asociados en eucariotas, destacando la función del ARN mensajero y las ARN polimerasas. Se describen las etapas de inicio, elongación y terminación de la transcripción, así como el procesamiento del ARN. También se discuten las diferencias entre procariotas y eucariotas y la interacción de factores de transcripción en la regulación de la expresión génica.