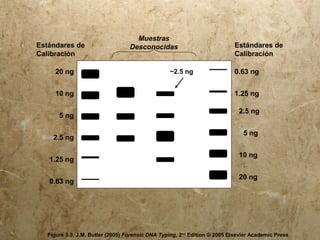
Este documento describe varios métodos para la extracción y cuantificación de ADN, incluyendo extracción orgánica, por Chelex, y usando papel FTA. También cubre temas como la cuantificación de ADN mediante espectrofotometría y fluorometría, y el uso de PCR en tiempo real (qPCR).