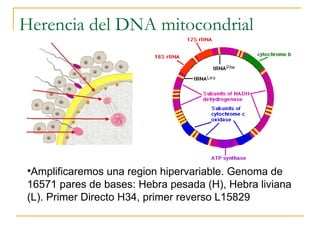
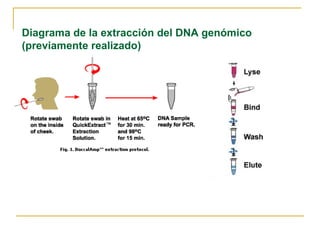
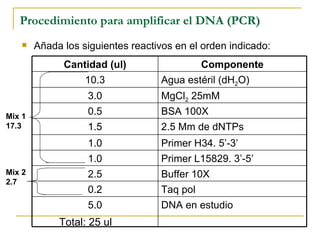
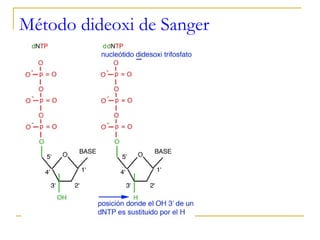
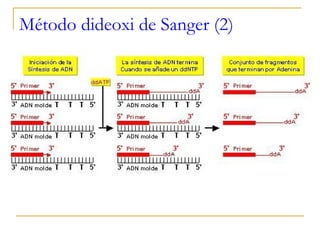
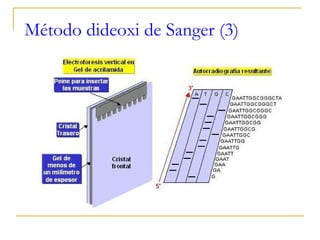
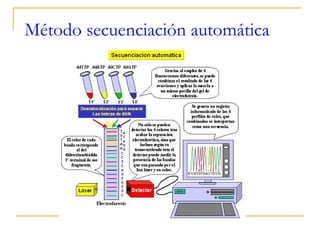
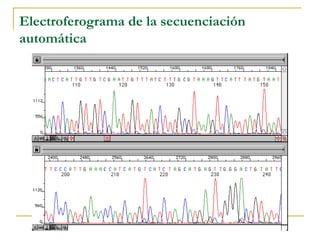
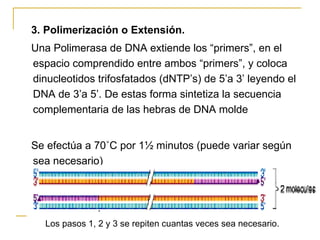
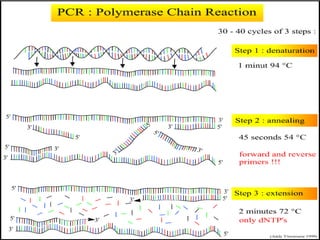
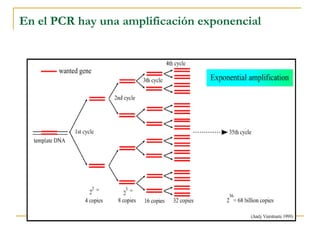
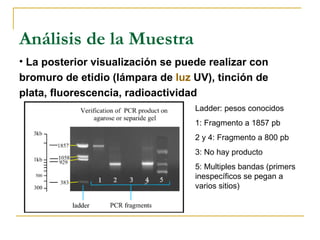
Este documento describe los fundamentos, pasos y aplicaciones de la reacción en cadena de la polimerasa (PCR), incluyendo la amplificación exponencial de ADN, los reactivos necesarios como cebadores, polimerasa y dNTPs, y los usos de PCR en análisis genéticos, investigación forense y medicina.















![Materiales Para PCR Termociclador “Thermo cycler” Microcentrífuga Micropipetas de 2, 20 y 200 ul Microtubos para “PCR”, estériles Puntas estériles Agua destilada, desionizada y estéril ADN molde (ADN en estudio) Primer Forward y Reverse dNTP’s ( dATP, dTTP, dCTP, dGTP de [25 μ M] c/u) 10X buffer para PCR ( Solución amortiguadora para “PCR”) MgCl 2 (25 mM) BSA Polimerasa Taq](https://image.slidesharecdn.com/pcr-100112155725-phpapp01/85/Pcr-23-320.jpg)