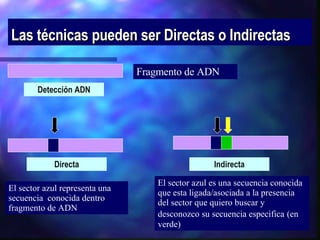
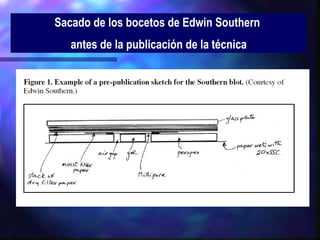
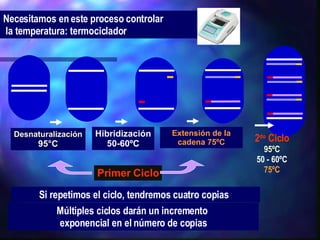
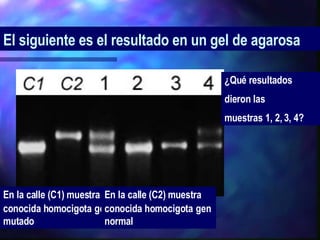
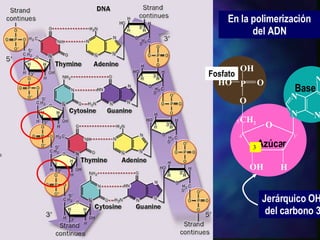
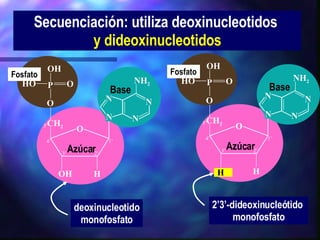
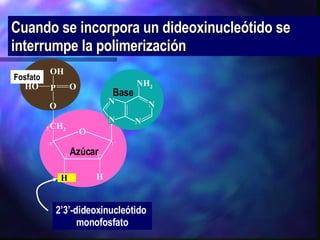
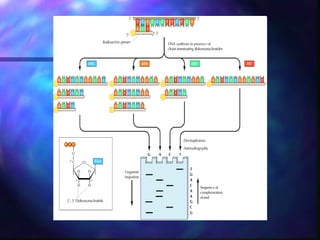
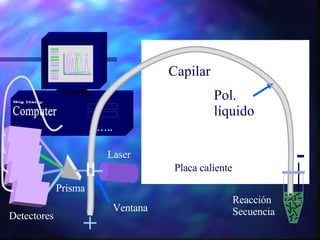
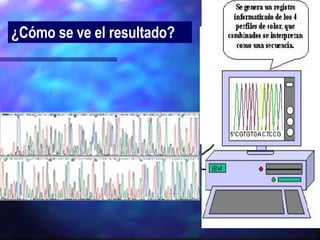
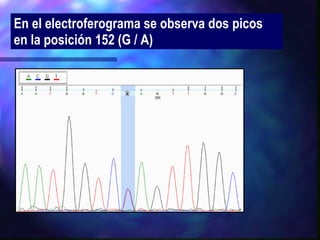
El documento resume diferentes técnicas de biología molecular utilizadas para extraer, almacenar y analizar ADN, incluyendo la extracción de ADN de muestras biológicas, el almacenamiento a bajas temperaturas, el Southern blot para detección de secuencias específicas, la reacción en cadena de la polimerasa (PCR) para amplificación de fragmentos, y la secuenciación del ADN mediante terminadores de cadena.