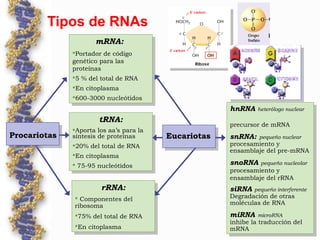
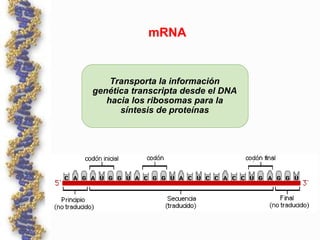
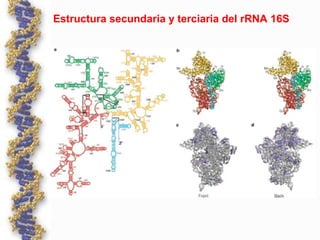
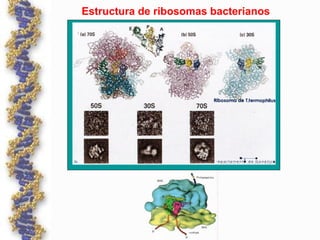
El documento describe los componentes y factores involucrados en el proceso de traducción en bacterias y eucariotas. Explica el papel del mRNA, tRNA, ribosomas y factores de traducción. Señala las diferencias entre los sistemas procariotas y eucariotas, como la presencia de factores múltiples en eucariotas y mRNA monocistrónico. También compara la estructura y función de estos componentes entre ambos dominios biológicos.