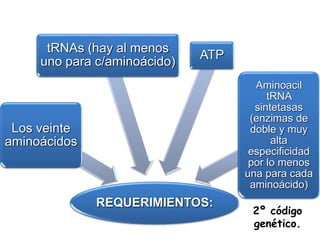
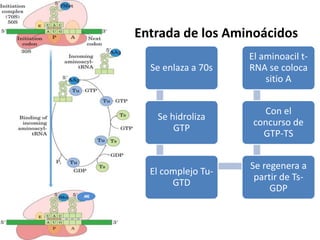
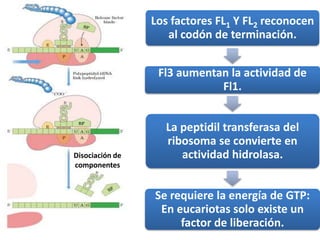
Este documento describe las etapas de la biosíntesis de proteínas. Explica que la traducción ocurre en los ribosomas y requiere ARN mensajero, ARN de transferencia y aminoácidos. Detalla las cuatro etapas principales: 1) activación de los aminoácidos, 2) iniciación, 3) elongación o alargamiento, y 4) terminación. También describe los roles del ARN mensajero, ARN de transferencia, ribosomas y otros factores en el proceso de traducción.