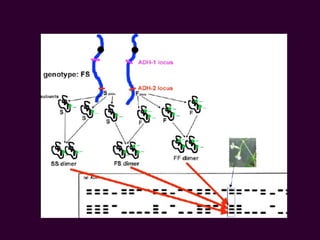
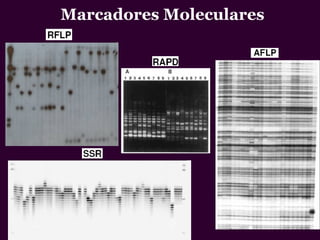
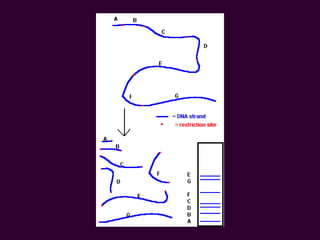
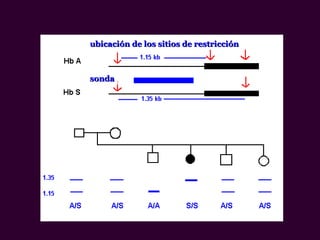
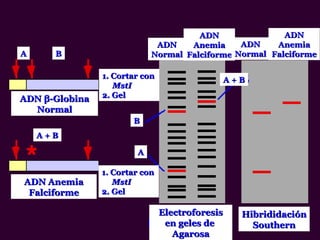
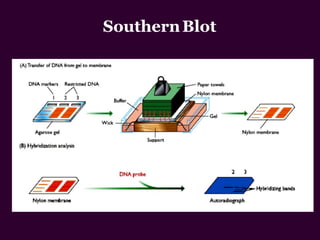
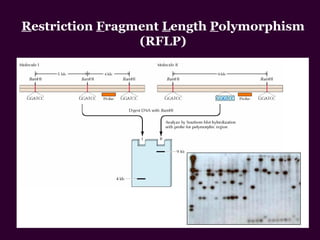
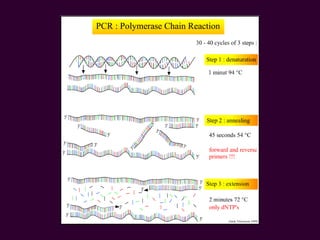
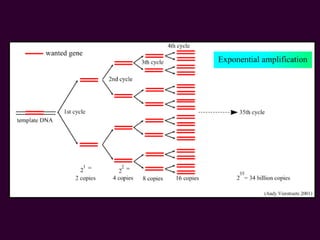
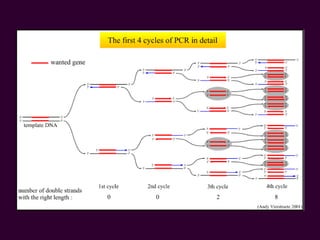
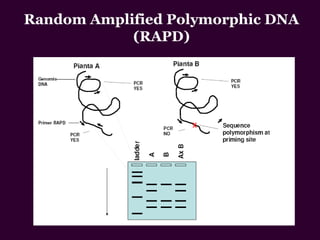
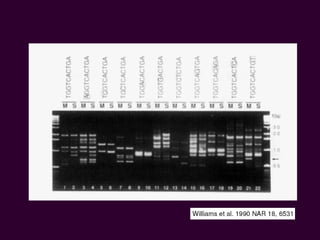
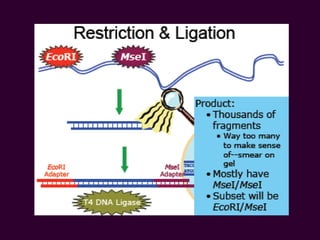
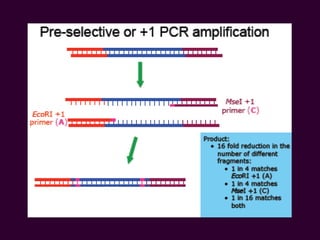
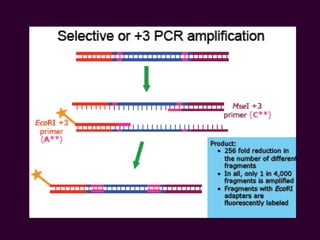
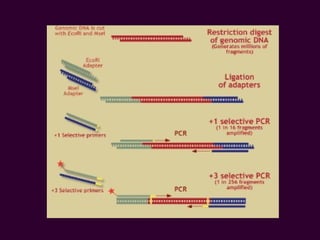
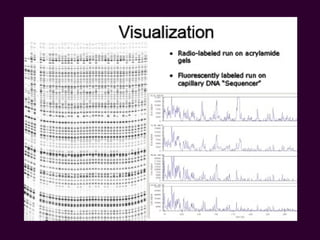
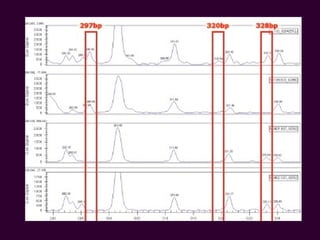
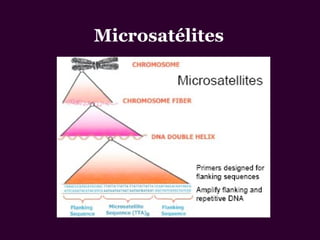
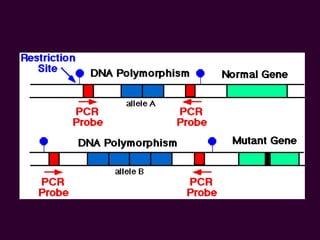
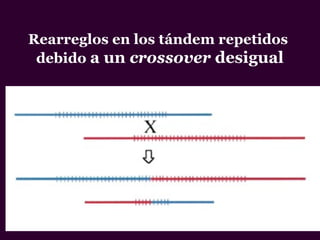
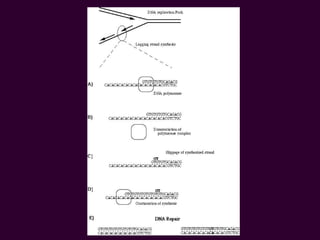
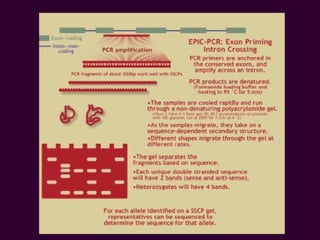
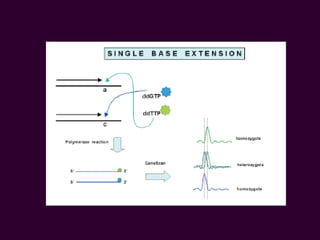
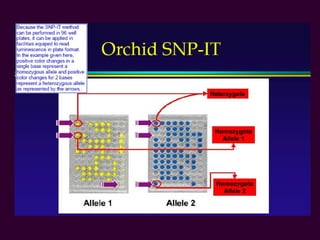
Marcadores moleculares son segmentos de ADN que pueden usarse para identificar organismos y características asociadas. Existen varios tipos de marcadores como RFLP, RAPD, AFLP, microsatélites y SNPs. Cada marcador tiene ventajas y desventajas dependiendo del propósito, aunque todos son mejores que marcadores morfológicos para mapeo genético.