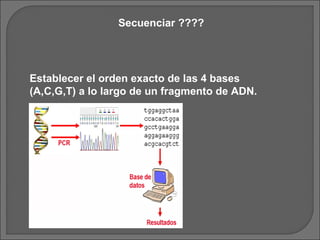
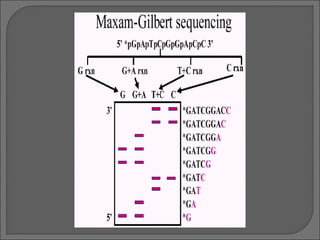
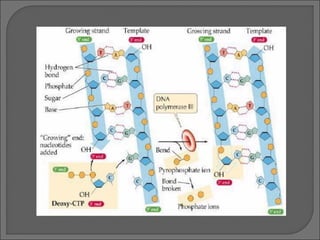
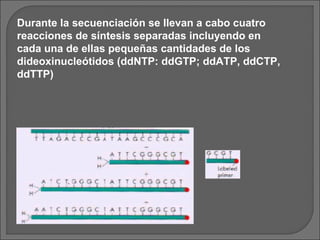
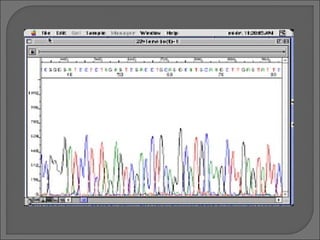
El documento describe los métodos de secuenciación de ADN desarrollados por Maxam-Gilbert y Sanger. Estos métodos permiten establecer el orden exacto de las bases A, C, G y T a lo largo de un fragmento de ADN y han sido fundamentales para secuenciar genomas completos y aplicaciones como el diagnóstico de enfermedades.