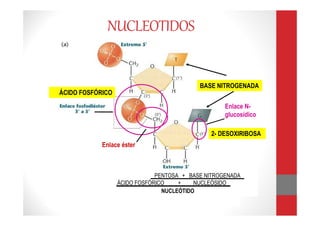
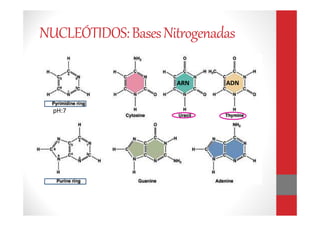
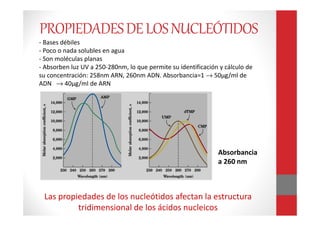
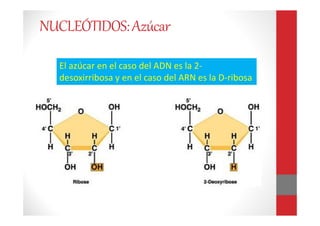
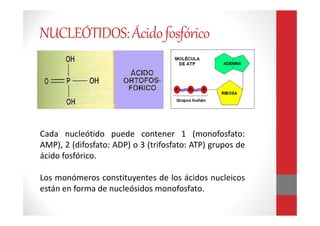
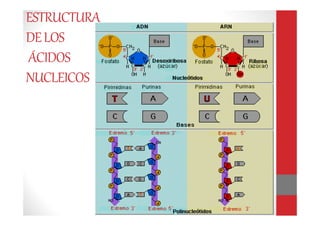
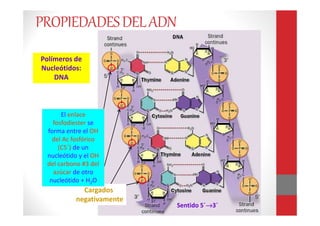
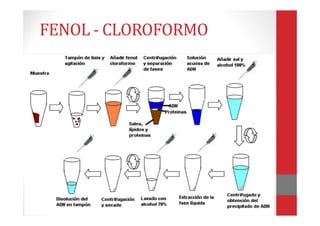
Este documento describe las técnicas de extracción de ácidos nucleicos como el ADN y el ARN. Brevemente explica que el ADN y el ARN son macromoléculas formadas por nucleótidos que contienen la información genética de los seres vivos. Luego resume los principales métodos de extracción como fenol-cloroformo, Chelex, papel FTA, columnas de sílica y salting-out, destacando sus ventajas y desventajas. Finalmente, brinda detalles sobre las muestras, factores que a











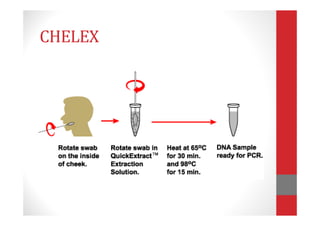
![CHELEX
• Técnica inorgánica.
• Es un copolímero quelante de iones metálicos como el Mg2+ inactiva
las DNasas y otras enzimas que pueden interferir en la PCR y otras
aplicaciones basadas en enzimas (ligación, etc).
• Elimina eficientemente los productos de la lisis celular (fuentes de
ADN: sangre total, bacterias y cultivo de células).
• Los reactivos no son tóxicos y no se pierde ADN.
• Pueden utilizarse en muestras con ↑[sales].
• Ventaja: económica, sencilla y rápida (un simple paso de ebullición en
presencia de la matriz, se utiliza el SN directo en la PCR).
DESVENTAJAS:
• ADN aislado es de cadena sencilla (desnaturalizado), utilizado en PCR
pero no en RFLPs.](https://image.slidesharecdn.com/extracciondeadn-190705053407/85/Extraccion-de-adn-21-320.jpg)


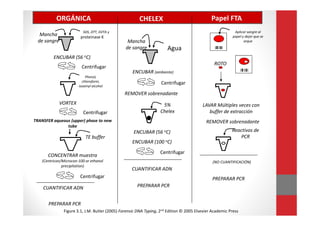

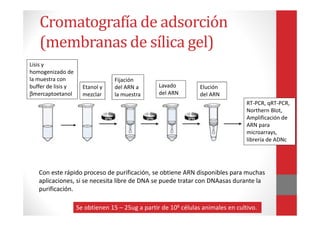
![• Bajo condiciones nativas, los Ac. nucleicos están recubiertos de una
capa de agua que mantienen su solubilidad en soluciones acuosas.
• Con la adición de iones caotrópicos, se destruye esta capa hidratante,
creando un entorno hidrofóbico, lo cual permite que el ADN se una a la
membrana de sílica de las columnas, mientras que las proteínas y otros
contaminantes no se unen y son eliminados durante los pasos de
lavado.
• Los ácidos nucleicos se eluyen de la membrana utilizando tampones de
elución con ↓ [sales] (ligeramente alcalinos) o agua, que permiten
recuperar la capa hidratante liberándolos de la membrana, sin afectar
los Ac. nucleicos.
Cromatografía de adsorción
(membranas de sílica gel)](https://image.slidesharecdn.com/extracciondeadn-190705053407/85/Extraccion-de-adn-27-320.jpg)