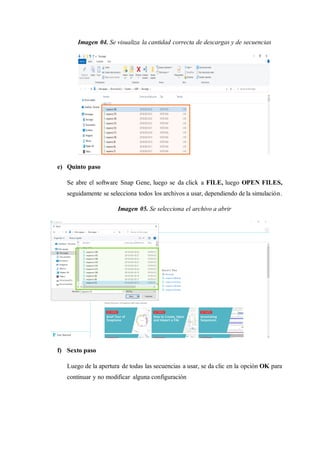
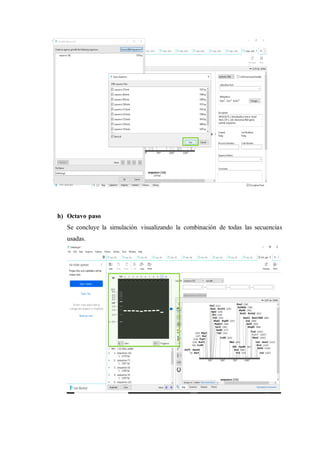
El documento describe la simulación de un gel de electroforesis usando el software SnapGene basado en un artículo científico. Explica brevemente el funcionamiento de la electroforesis y cómo separa moléculas como ADN basado en su tamaño. Luego detalla los pasos para realizar la simulación en SnapGene incluyendo buscar secuencias en NCBI y modelar los resultados en el software.