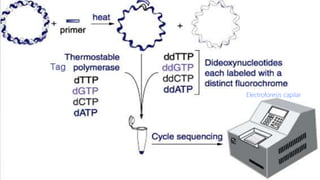
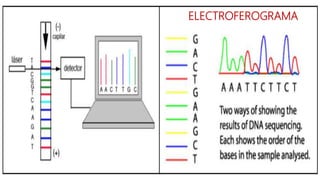
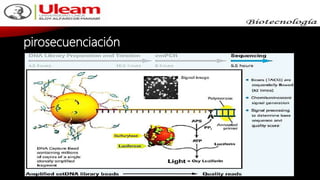
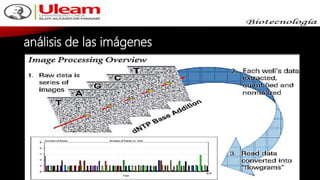
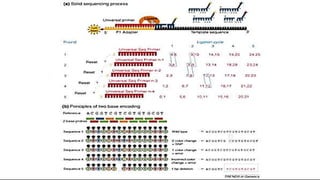
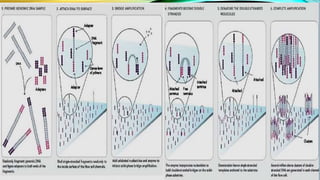
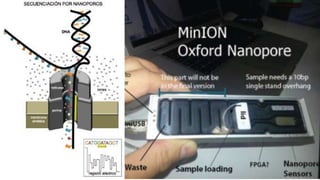
Este documento describe los diferentes métodos de secuenciación de ADN, incluyendo los métodos de primera generación como Sanger y pirosecuenciación, los métodos de segunda generación como secuenciación por síntesis utilizada por Illumina, y los métodos de tercera generación como secuenciación en tiempo real de Pacific Biosciences y Oxford Nanopore. Explica los principios, procedimientos y aplicaciones de cada método para determinar el orden de las bases nucleotídicas en una molécula de ADN.